Sección 5 Visualización de datos
La visualización de datos es una habilidad fundamental para el análisis de datos, tanto para comunicar los resultados como para un análisis exploratorio de datos. R cuenta con varios paquetes para realizar gráficos, por ejemplo graphics, ggplot2, plottly.
5.1 Graphics
Es el paquete base de R para realizar plots, ya está instalado por default en R. La función principal es plot y se puede usar con varias otras funciones de análisis en R.
A la función plot, podemos indicarle uno o dos vectores a graficar.
Estos son los scatterplot básicos, si son dos vectores se interpretan como coordenadas \(X,Y\), en el caso de un vector este se graficará en la coordenada Y y sobre la coordenada \(X\) se graficara en los puntos 1:n.
Esta función plot tiene muchos argumentos que podemos especificar como color, línea, texto. Explora la ayuda de R de la función plot.
Algunos de los argumentos de la función plot son los siguientes:
pch: cambia el símbolo por default de punto por algún otro símbolo. Para cambiar el parámetro usamospch=n. Algunos de los símbolos que se pueden usar son los siguientes.

cex: rescala el tamaño de los puntos. Por default el valor es1.col: cambia el color de los puntos. Admite nombre de colores comocol="red"o códigos comocol=2. El default escol=1. Algunos de los colores disponibles son los siguientes:
## [1] "white" "aliceblue" "antiquewhite" "antiquewhite1"
## [5] "antiquewhite2" "antiquewhite3" "antiquewhite4" "aquamarine"
## [9] "aquamarine1" "aquamarine2" "aquamarine3" "aquamarine4"
## [13] "azure" "azure1" "azure2" "azure3"
## [17] "azure4" "beige" "bisque" "bisque1"## Loading required package: grid
También se dispone del paquete RColorBrewer.

lty: cambia el tipo de línea de sólida a punteada. Los valores van de 0 a 6.
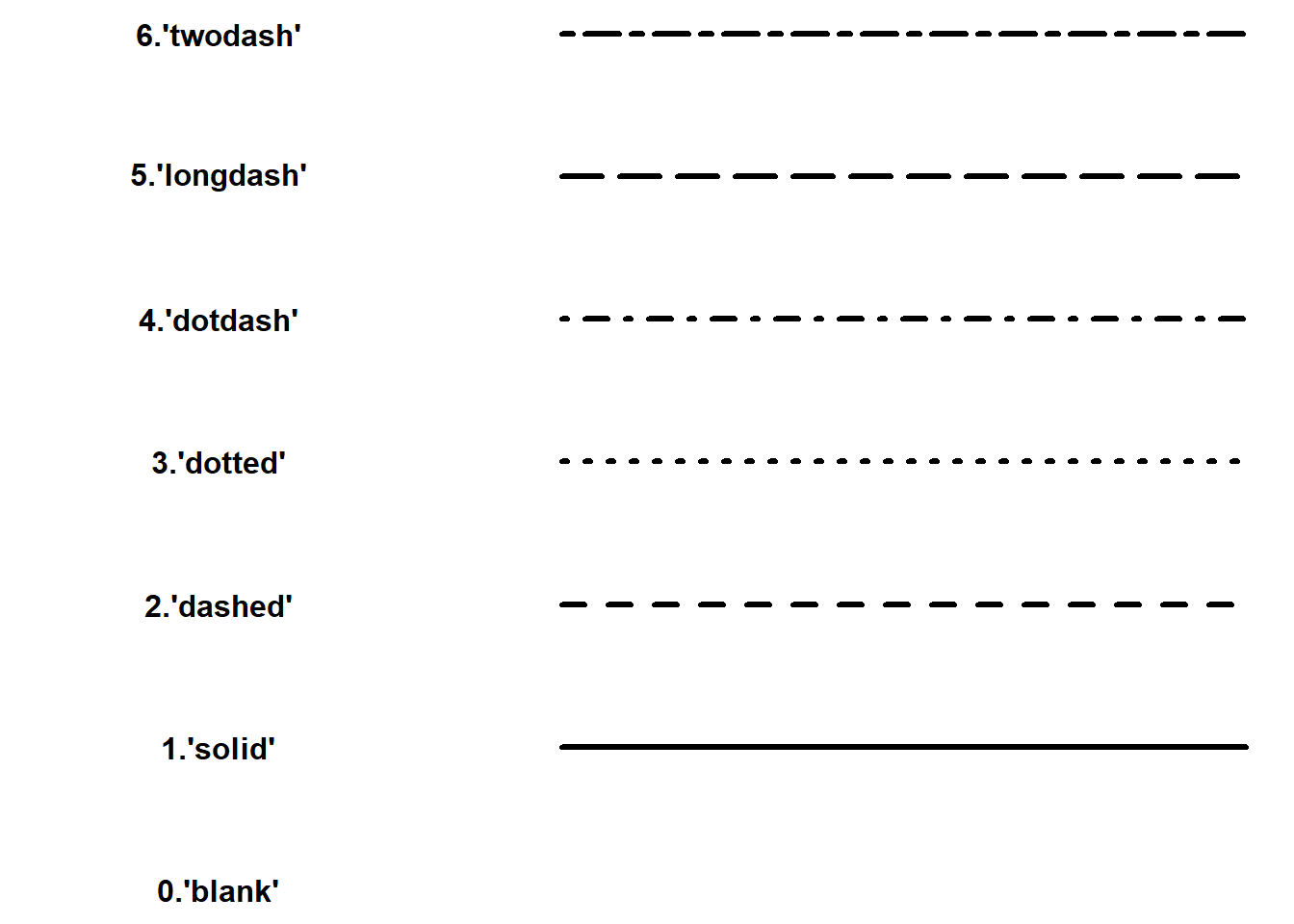
lwd: cambia el grosor de la línea. Por default eslwd=1.main: se usa para cambiar el título del plot.xlab,ylab: sirven para cambiar las etiquetas de los ejes \(xX,Y\).xlim,ylim: cambia los límites de los ejes.type: sirve para cambiar el tipo de plot que vamos a usar.type:"p": para puntos, es el valor default.type:"l": para líneas.type:"b": para puntos conectados por líneas.type:"o": para puntos conectados por líneas sin dejar espacios.type:"h": para histogramas, es decir líneas verticales.type:"s": para como escalera.type:"n": para no mostrar nada.
axes: es un parámetro lógico para remover los ejes.bty="n": para remover el grid.
En ocasiones queremos desplegar varias gráficas en un arreglo \(n\times m\). Para realizar esto usamos la función siguiente
Esta configuración va a continuar hasta que se indique lo contrario.
par(mfrow=c(2,2))
x <- 1:10
y <- x*x
plot(x,y, type="b", lwd=2)
plot(x,y, pch=7, cex=0.9, col="aquamarine3", main = "y=x*x",
xlab="Eje X", ylab= "Eje Y")
plot(x,y, lty= 2, col=3, type = "b", cex=0.8, xlab="", ylab="", xlim = c(0,15) )
plot(x,y, pch=25, cex=2, col="purple1", axes=FALSE, bty="n", bg="pink")
5.1.1 Scatterplot: gráficos de dispersión
La función plot también puede recibir bases de datos o matrices. Si el argumento que se le pasa es una matriz, entonces solo usará las primeras dos columnas y si por el contrario es un data.frame entonces graficará varios plots con todas las posibles combinaciones 2 a 2 de variables.
Vamos a cargar la base de datos iris.
## 'data.frame': 150 obs. of 5 variables:
## $ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
## $ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
## $ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
## $ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
## $ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...
Los mismos argumentos de la primera sección se le puede aplicar a estos gráficos.
plot(iris$Sepal.Length, iris$Sepal.Width, pch=17, cex=0.7, col=iris$Species,
main="Sepal length and width", xlab="Sepal Length", ylab="Sepal Width")
En el gráfico no sabemos que color corresponde a que especie. Podemos agregar legendas o cuadros de texto para indicar esto.
plot(iris$Sepal.Length, iris$Sepal.Width, pch=17, cex=0.7, col=iris$Species,
main="Sepal length and width", xlab="Sepal Length", ylab="Sepal Width")
legend("bottomright", legend = levels(iris$Species), col = 1:3, pch = 17, pt.cex = 0.9)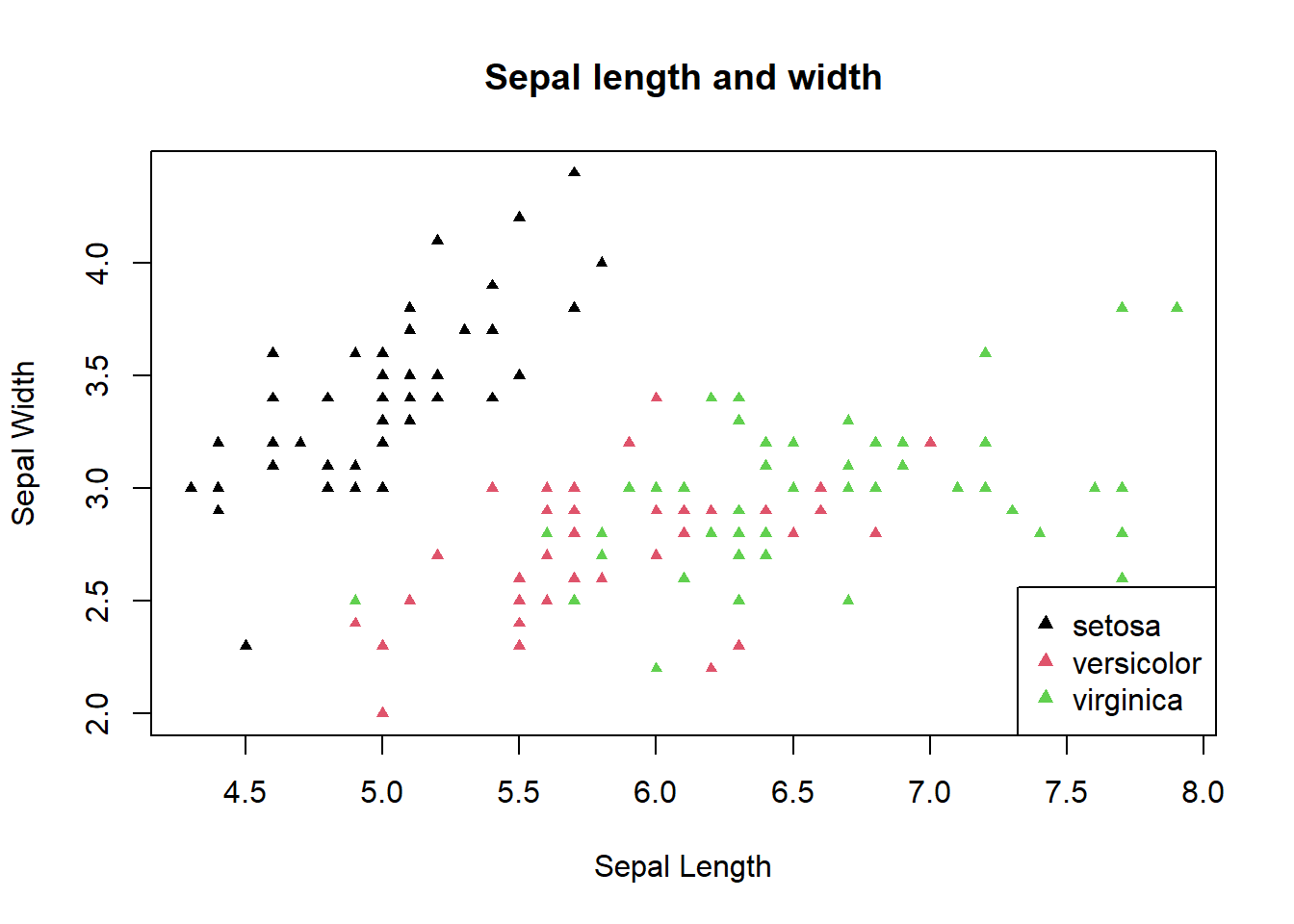
Usando las paletas de colores de RColorBrewer:
cl <- brewer.pal(n=3, name="Set2")
plot(iris$Sepal.Length, iris$Sepal.Width, pch=17, cex=0.7, col=cl[iris$Species],
main="Sepal length and width", xlab="Sepal Length", ylab="Sepal Width")
legend("bottomright", legend = levels(iris$Species), col = cl[1:3], pch = 17, pt.cex = 0.9)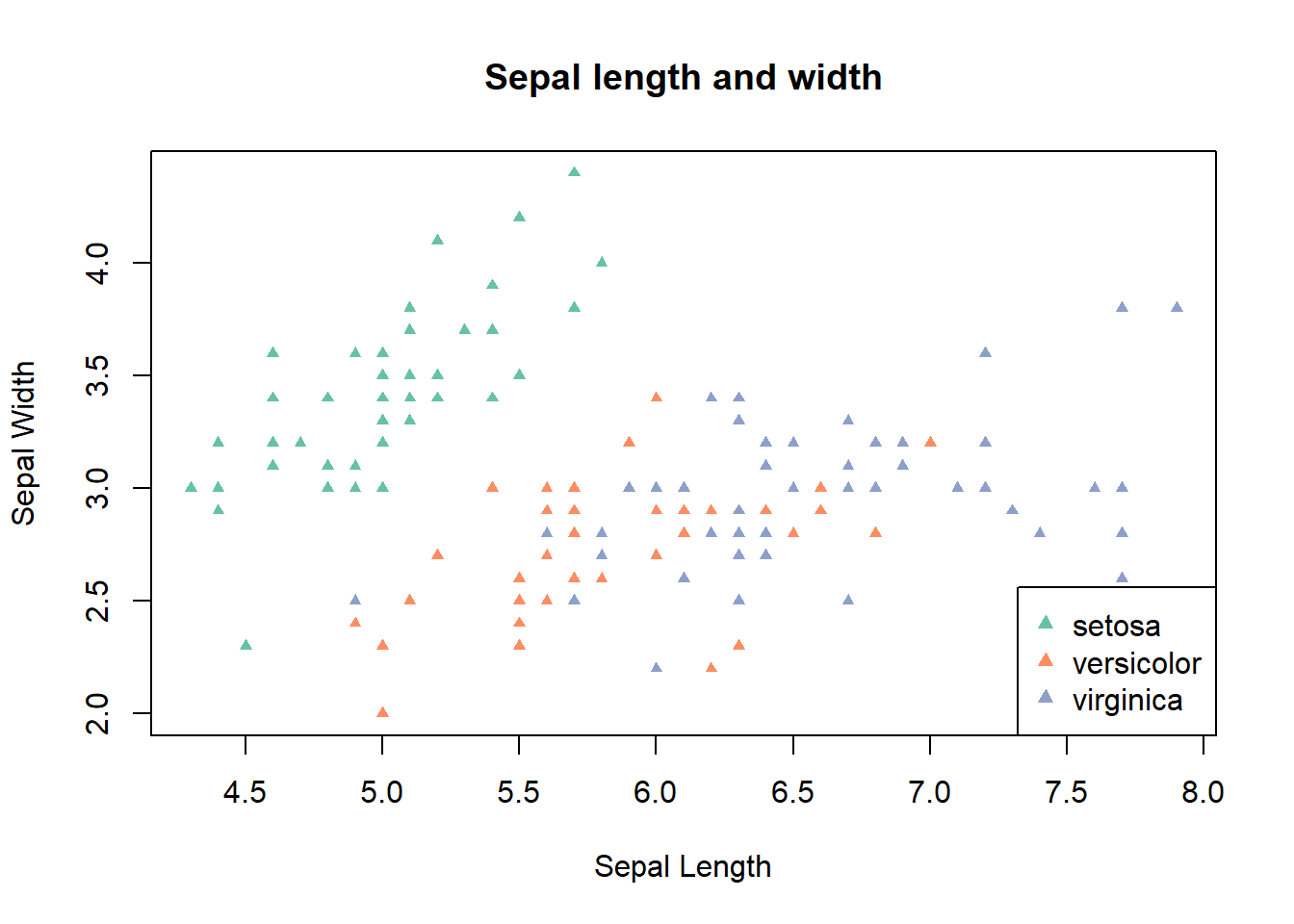
Especificando colores con su formato hexadecimal.
cl <- c("#FF33CC", "#CC66FF", "#33C9CC")
plot(iris$Sepal.Length, iris$Sepal.Width, pch=17, cex=0.7, col=cl[iris$Species],
main="Sepal length and width", xlab="Sepal Length", ylab="Sepal Width")
legend("bottomright", legend = levels(iris$Species), col = cl[1:3], pch = 17, pt.cex = 0.9)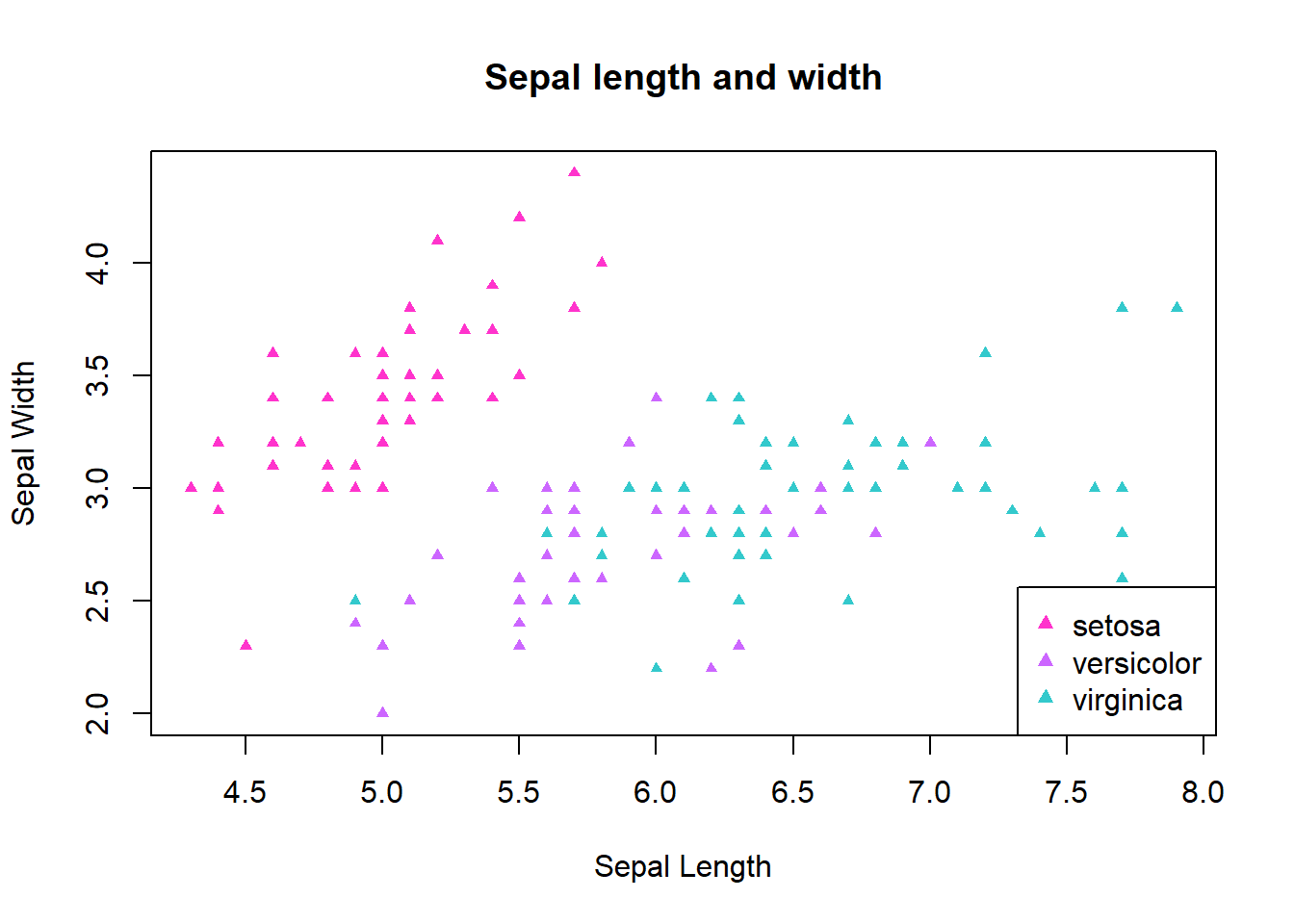
Nota: Links a varias paletas de colores:
- library(paletter)
- library(RColorBrewer)
- Palette generator
- Formato hexadecimal
Otros gráficos de dispersión en arreglo de matrices se pueden obtener con la función pairs.
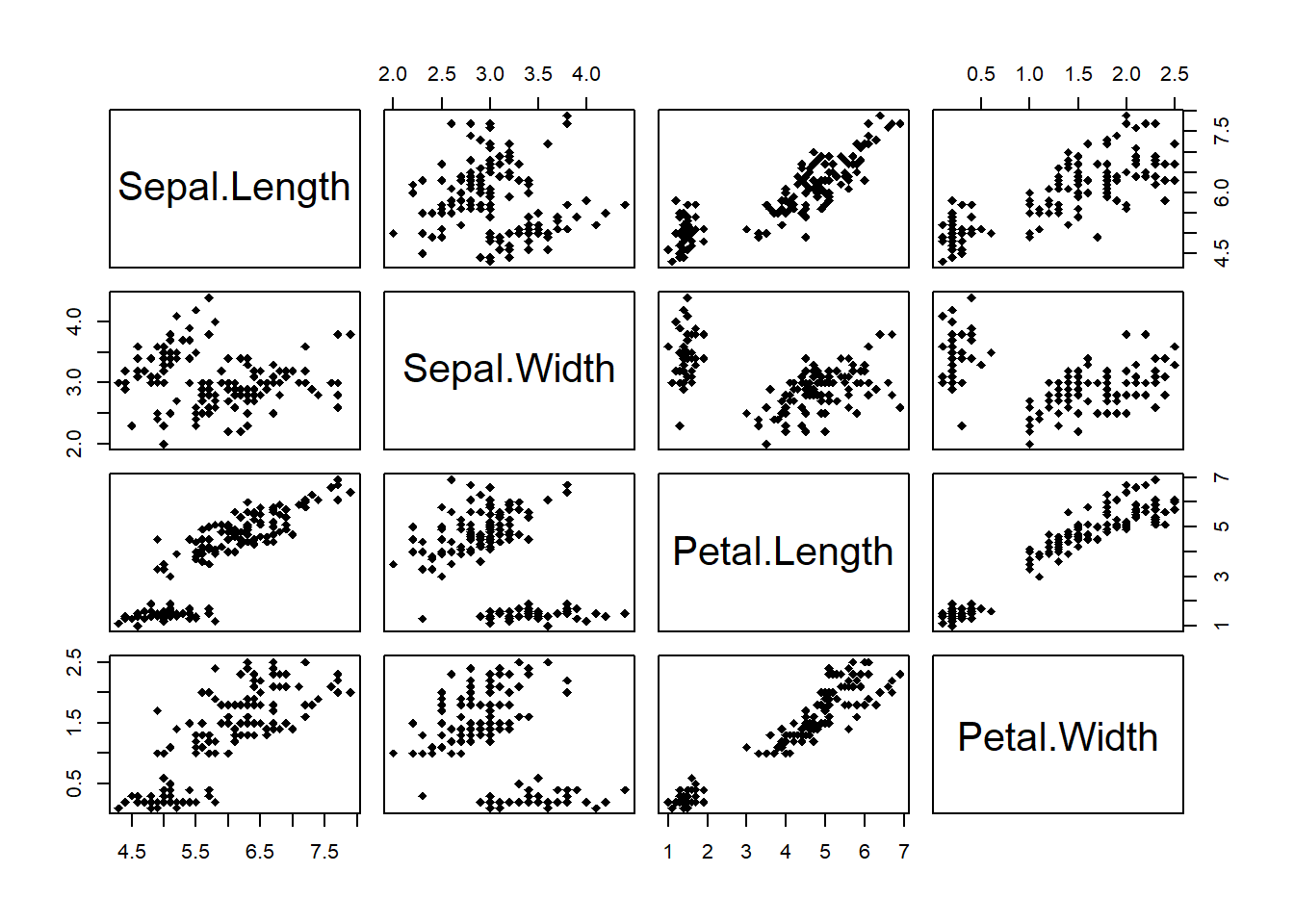
Al igual que con los anteriores, podemos configurar varias opciones como colores, si queremos que aparezca solo la diagonal superior, el tipo, legendas, etc.
cl <- brewer.pal(n=3, name="Dark2")
pairs(iris[,1:4], pch=17, cex=0.5, col=cl[iris$Species],
main="Scatterplot Iris", lower.panel = NULL, oma=c(3,3,3,15))
par(xpd = TRUE)
legend("bottomright", legend = c(levels(iris$Species)), pch=17, col=cl[1:3], pt.cex=1.2)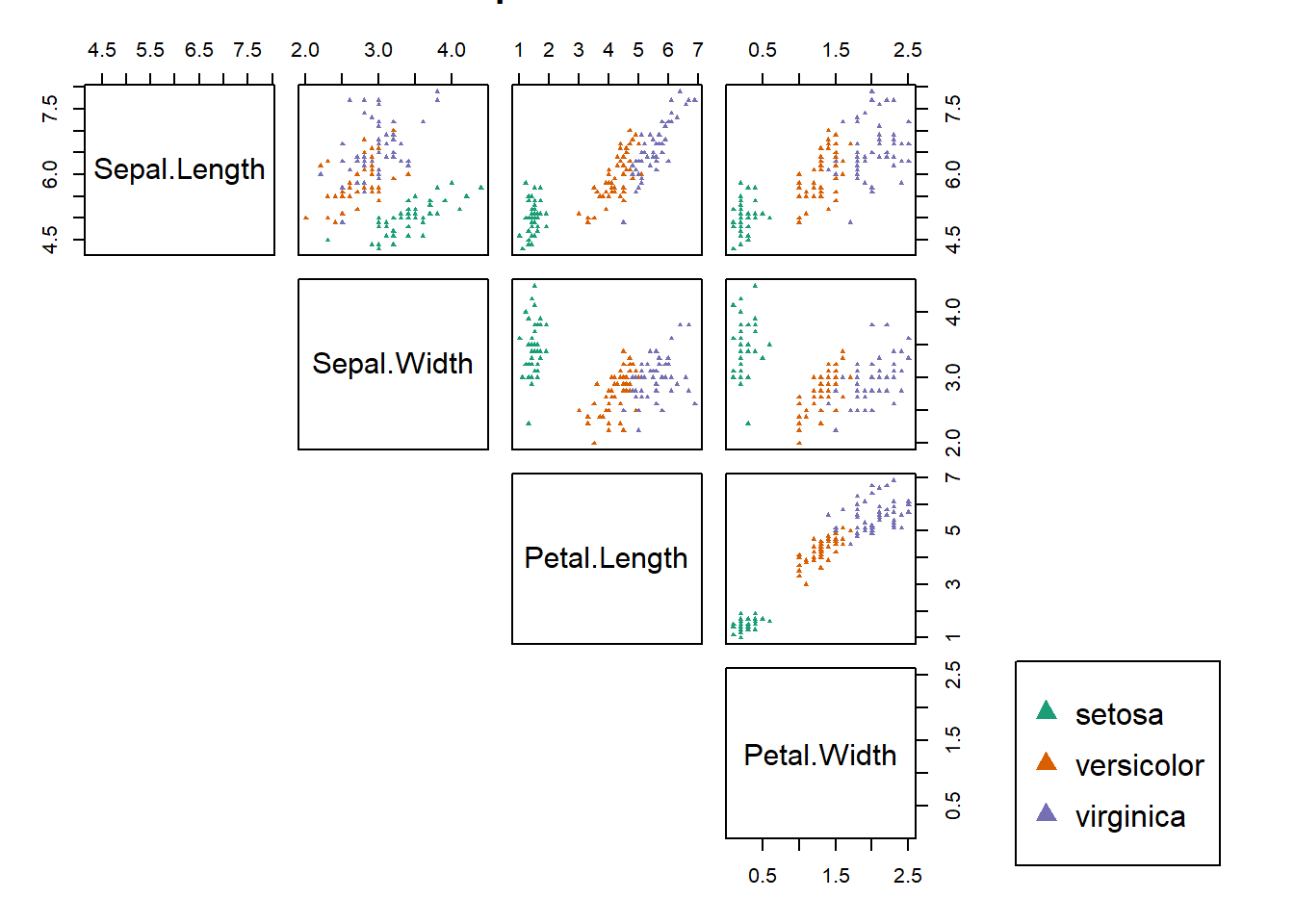
En este tipo de gráficos se pueden añadir por ejemplo los valores de las correlaciones, histogramas en la diagonal, densidades. Una forma de hacer esto es programando las funciones de lo que queremos que aparezca en panel superior, diagonal o inferior o usando la librería psych.
#install.packages("psych)
library(psych)
pairs.panels(iris[1:4], pch=21+as.numeric(iris$Species), bg=c("red","yellow","blue")[iris$Species],
oma=c(3,3,3,15),
method = "pearson", #método para la correlacion: pearson, spearman, kendall
hist.col = "#0099FF", # color a usar en los histogramas de la diagonal
density = TRUE, # valor lógico para mostrar los plots de densidad
cex.cor = 0.8 # modificar el tamaño de los números de correlación
)
par(xpd = TRUE)
legend("bottomright", legend = c(levels(iris$Species)), col=c("red","yellow","blue"), fill = c("red","yellow","blue"))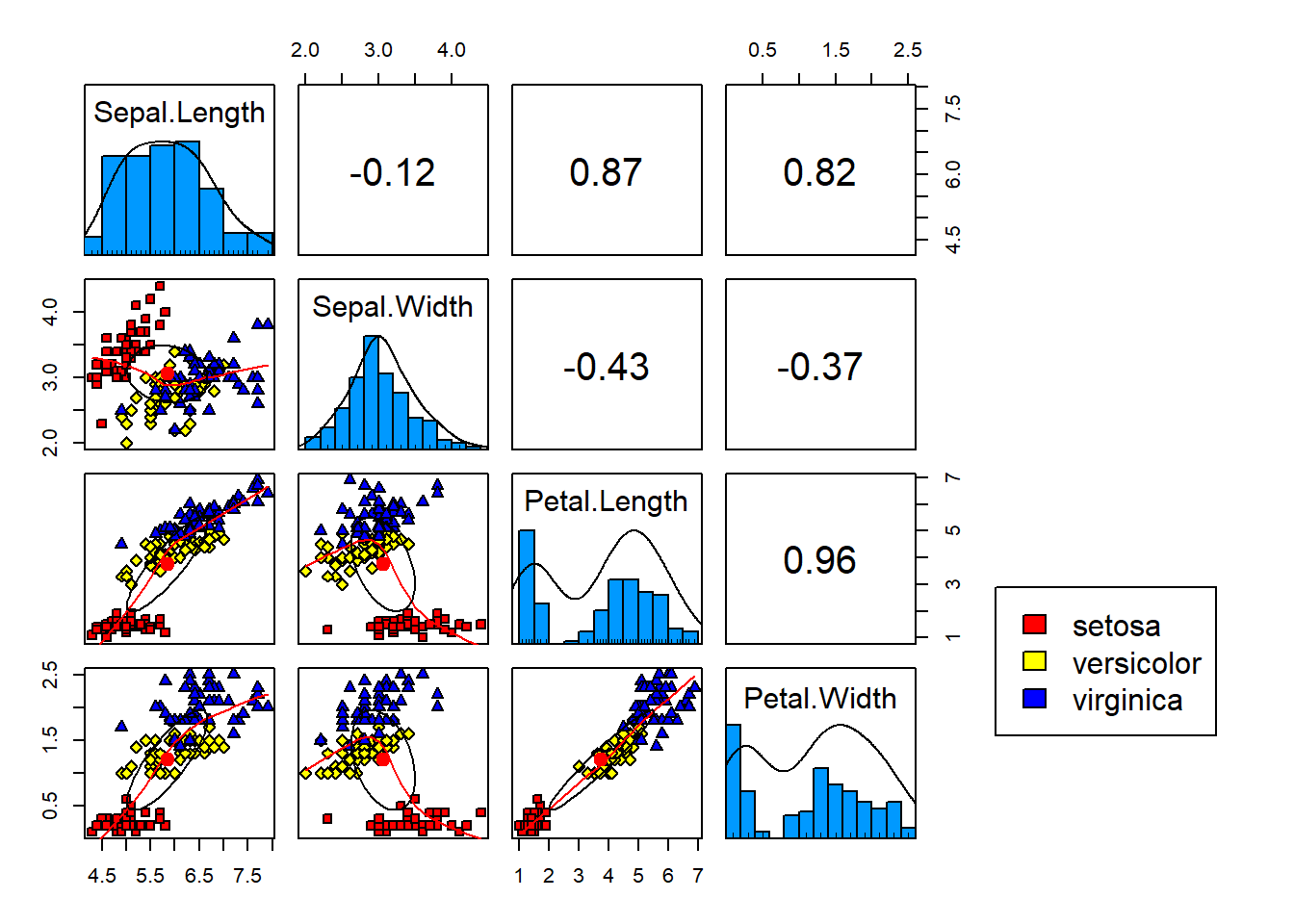
Un ejemplo de como usar funciones para realizar este plot es el siguiente, ver.
cl <- c("#FF33CC", "#CC66FF", "#33C9CC")
# Panel de correlación
panel.cor <- function(x, y){
usr <- par("usr"); on.exit(par(usr))
par(usr = c(0, 1, 0, 1))
r <- round(cor(x, y), digits=2)
txt <- paste0("R = ", r)
cex.cor <- 0.8/strwidth(txt)
text(0.5, 0.5, txt, cex = cex.cor * r)
}
# Panel superior
upper.panel<-function(x, y){
points(x,y, pch = 18, col = cl[iris$Species])
}
# Crear el plot
pairs(iris[,1:4], oma=c(3,3,3,15),
lower.panel = panel.cor,
upper.panel = upper.panel)
par(xpd = TRUE)
legend("bottomright", legend = c(levels(iris$Species)), pch=18, col=cl[1:3], pt.cex=1.2)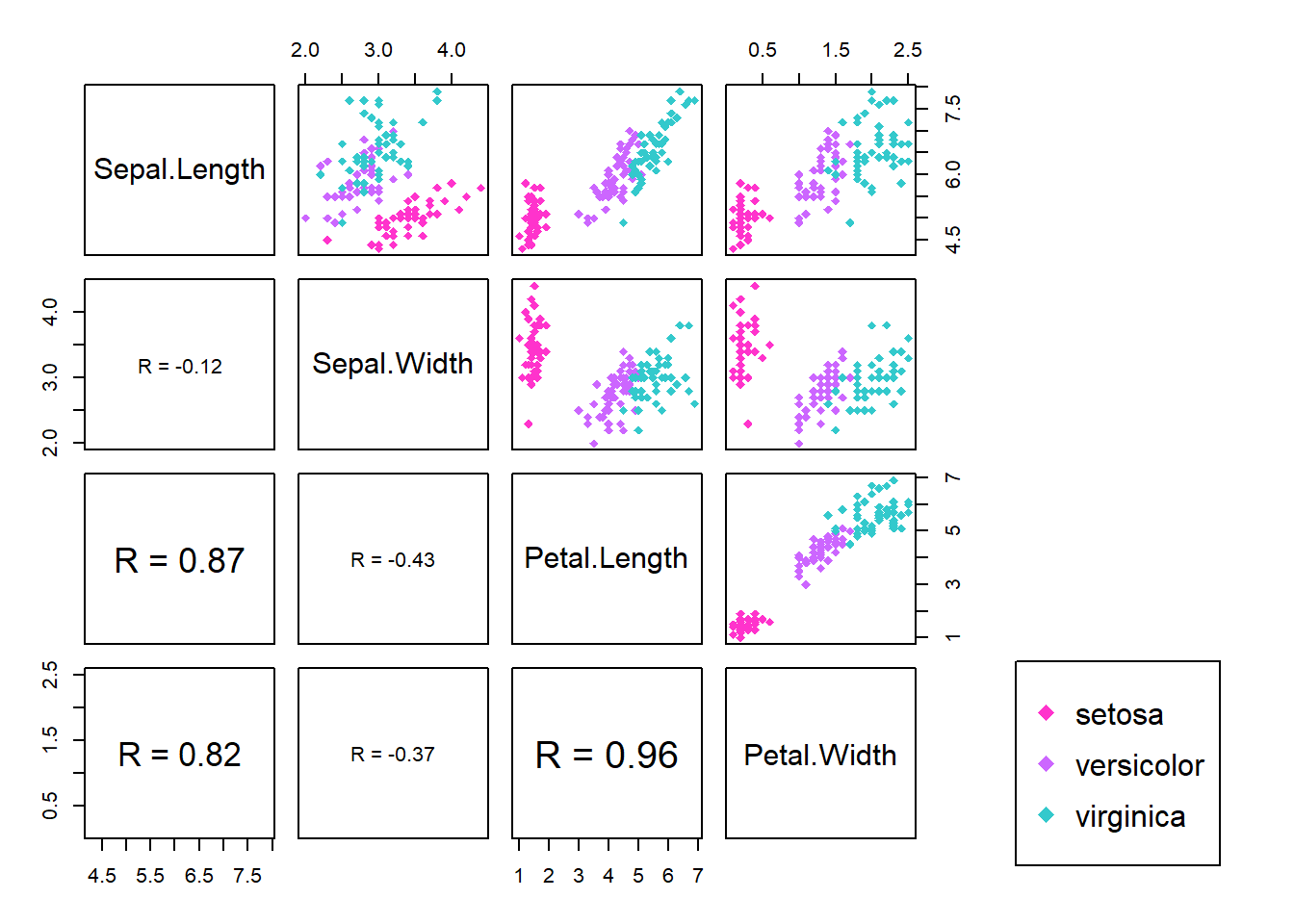
5.1.2 Barplot
Los gráficos de barras comparan valores en una serie de barras de la altura dada. La función que usamos del paquete base de R es barplot, esta toma como vector numérico y realiza una serie de barras de las alturas correspondientes.
Vamos a cargar la base de datos VADeaths del paquete datasets.
## Rural Male Rural Female Urban Male Urban Female
## 50-54 11.7 8.7 15.4 8.4
## 55-59 18.1 11.7 24.3 13.6
## 60-64 26.9 20.3 37.0 19.3
## 65-69 41.0 30.9 54.6 35.1
## 70-74 66.0 54.3 71.1 50.0El gráfico base se realiza indicando solamente la variable a graficar.
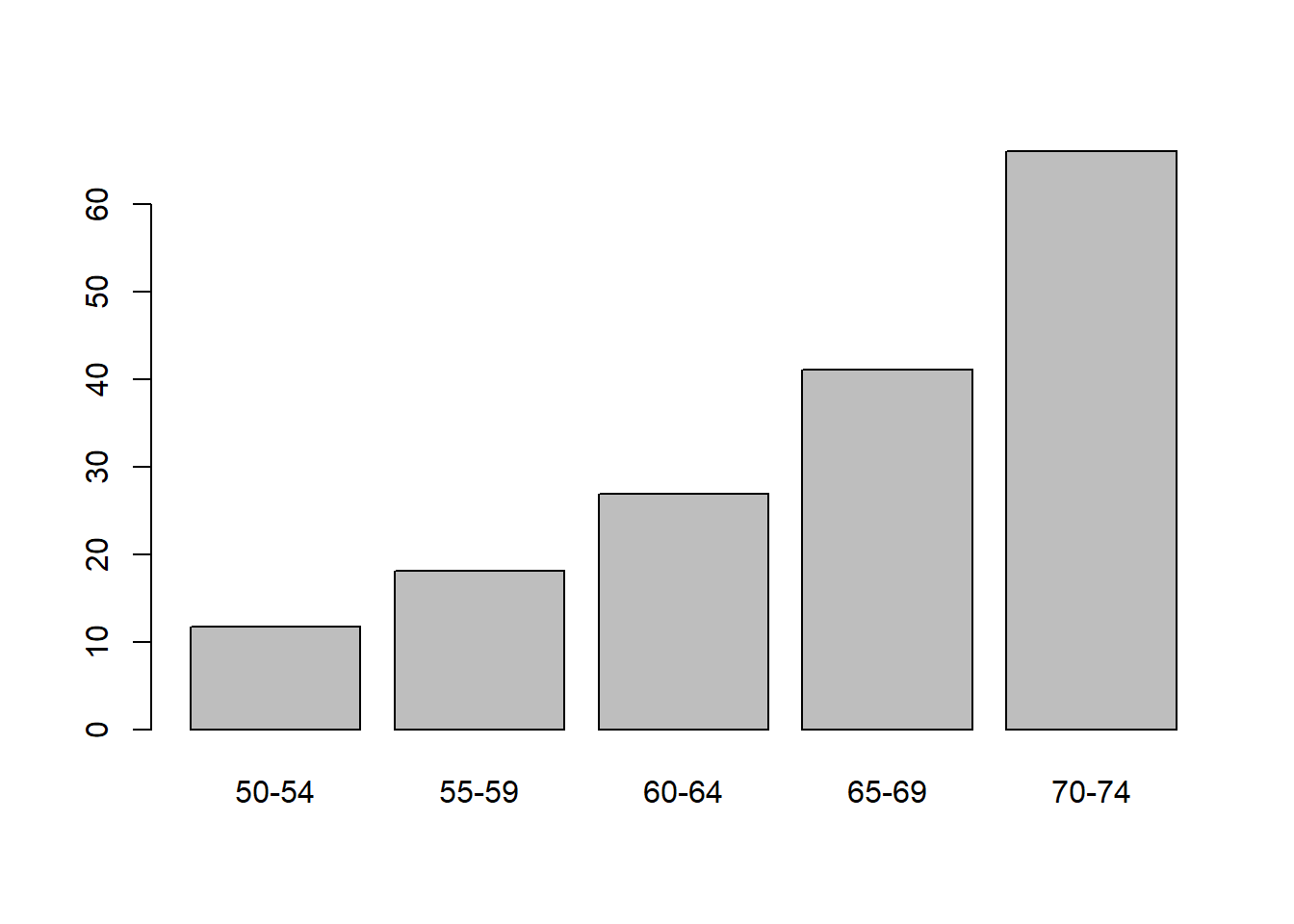
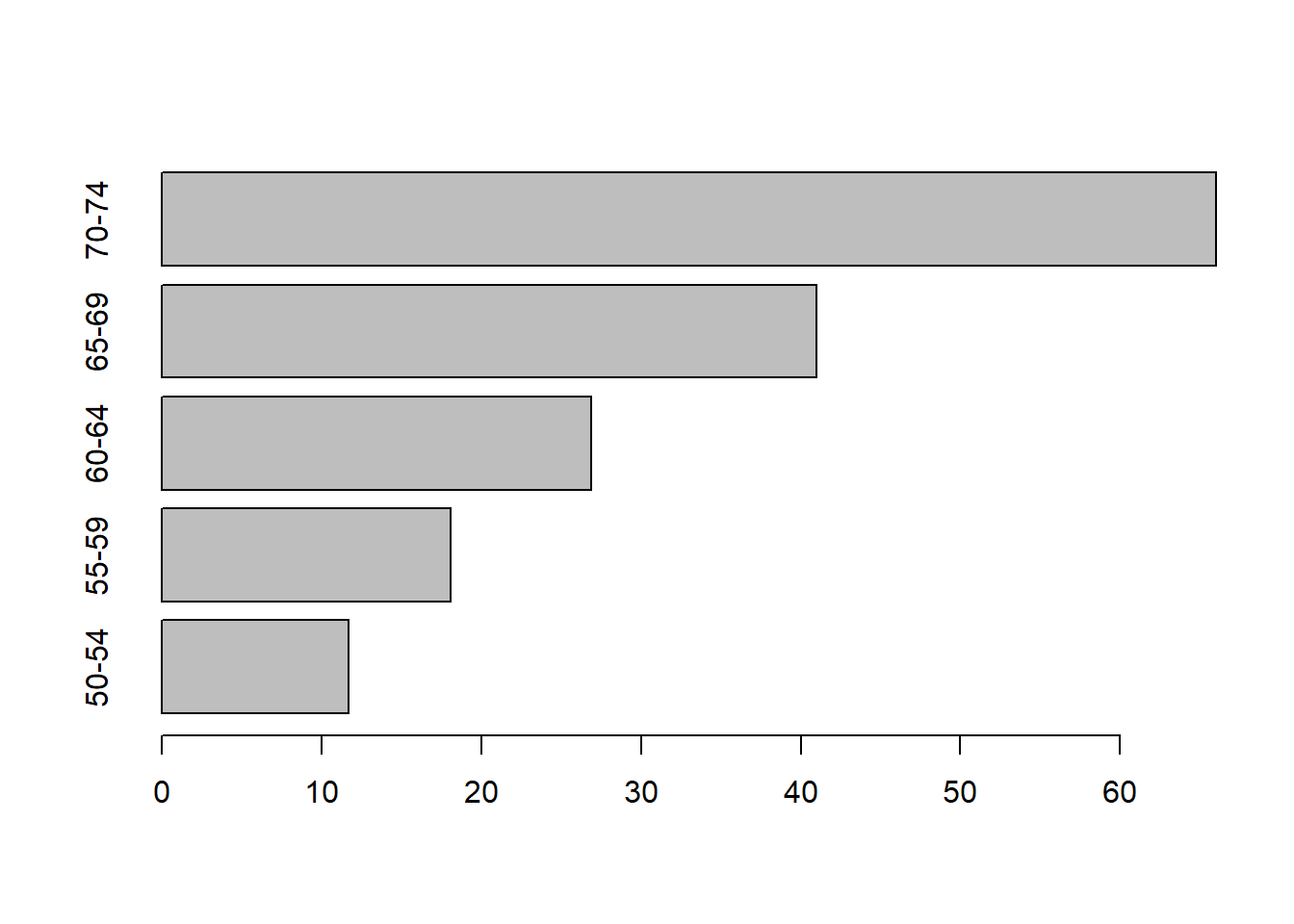
Al igual que los scatterplots podemos también cambiar colores, forma de agrupar, llenado, legendas, etc.
par(mfrow=c(2,2))
#Cambiar relleno y color de borde
barplot(VADeaths[,"Rural Male"], col = "white", border = "blue")
# Cambiar color de borde a cada barra
cl <- brewer.pal(n=9, name="Dark2")
barplot(VADeaths[,"Rural Male"], col = "white", border = cl[1:5])
# Cambiar color de relleno
barplot(VADeaths[,"Rural Male"], col = cl[1:5], border = "black")
# Cambiar título y nombre en ejes
barplot(VADeaths[,"Rural Male"], col = cl[1:5], border = "black",
main= "Taza de muertes en Virginia",
xlab = "Edad", ylab = "Taza")
# Añadir etiquetas de texto con el valor de la barra
p.barras <- barplot(VADeaths[,"Rural Male"], col = cl[1:5], border = "black", ylim = c(0,72))
text(p.barras, VADeaths[,"Rural Male"]+3.5, labels= VADeaths[,"Rural Male"])
# Añadir el grid
p.barras <- barplot(VADeaths[,"Rural Male"], col = cl[1:5], border = "black", ylim = c(0,70))
grid(nx=NA, ny=NULL, lwd = 1, lty = 1, col = "gray")
barplot(VADeaths[,"Rural Male"], col = cl[1:5], border = "black", ylim = c(0,70), add = TRUE)
# Cambiar las etiquetas de los grupos
p.barras <- barplot(VADeaths[,"Rural Male"], col = cl[1:5], border = "black",
ylim = c(0,70), names.arg = c("G1", "G2", "G3", "G4", "G5"))
par(mfrow=c(1,2))
# Cambiar el ancho de las barras
barplot(VADeaths[,"Rural Male"], col = cl[1:5], border = "black", ylim = c(0,70),
width = c(0.5,0.8,1.1,1.5,2))
# Cambiar el espacio entre las barras
barplot(VADeaths[,"Rural Male"], col = cl[1:5], border = "black", ylim = c(0,70),
space = c(0.5,0.8,1.1,1.5,2))
# Caja con leyendas
barplot(VADeaths[,"Rural Male"], col = cl[1:5], border = "black", ylim = c(0,70),
legend.text = rownames(VADeaths))
# Mover la caja de leyendas
barplot(VADeaths[,"Rural Male"], col = cl[1:5], border = "black", ylim = c(0,70),
legend.text = rownames(VADeaths),
args.legend = list(x="top"))
# Usando la función `legend`
barplot(VADeaths[,"Rural Male"], col = cl[1:5], border = "black", ylim = c(0,70))
legend("top", legend = rownames(VADeaths), fill = cl[1:5])
# Mover la caja de leyedas fuera del plot
barplot(VADeaths[,"Rural Male"], col = cl[1:5], border = "black", xlim=c(0,7.5),
ylim = c(0,70),
legend.text = rownames(VADeaths), beside = TRUE
#args.legend = list(x="topright", inset=c(-0.10,0))
)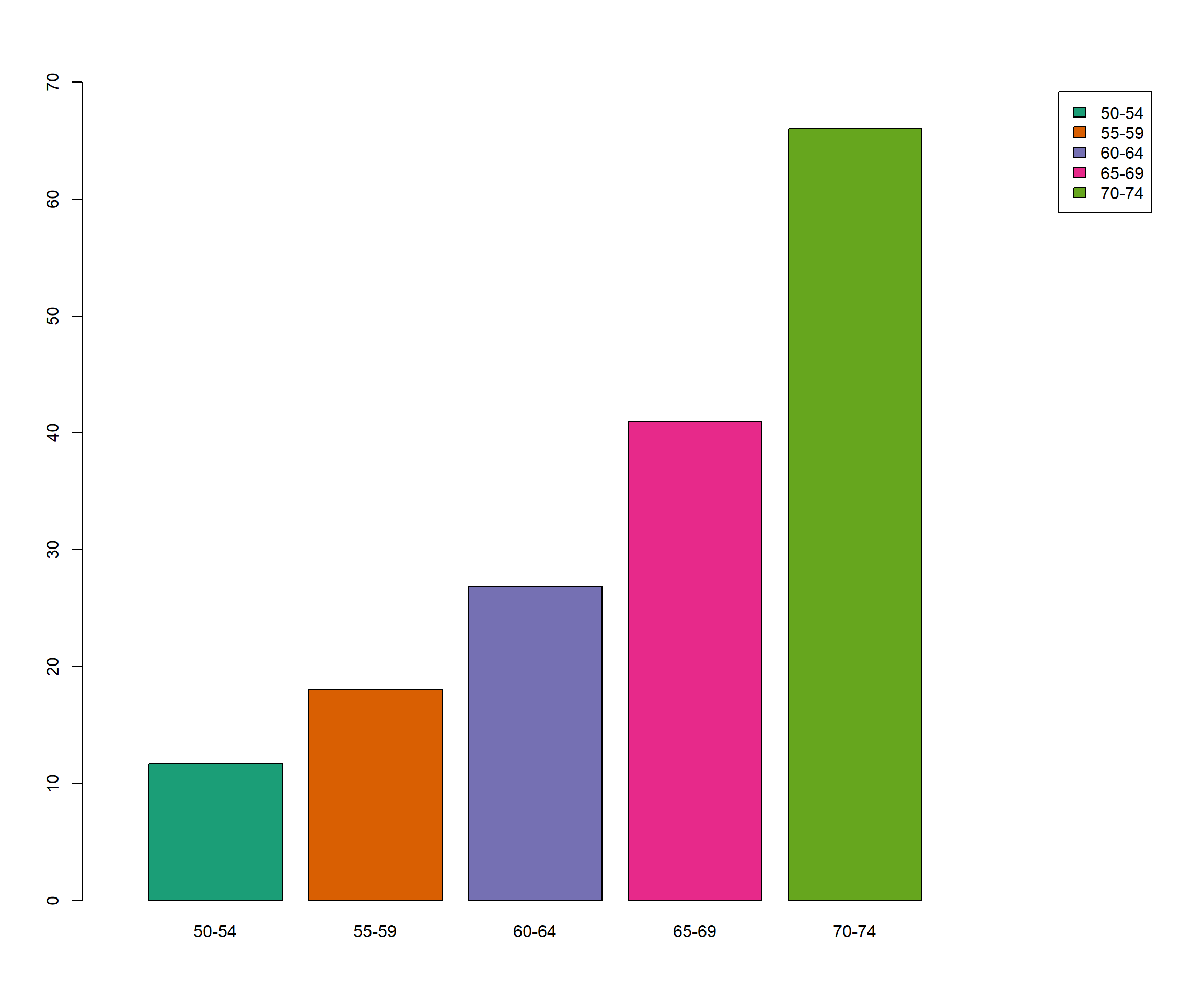
# Barras apiladas
barplot(VADeaths, col = cl[1:5], border = "black", xlim=c(0,6.5), cex.names = 0.7,
legend = rownames(VADeaths))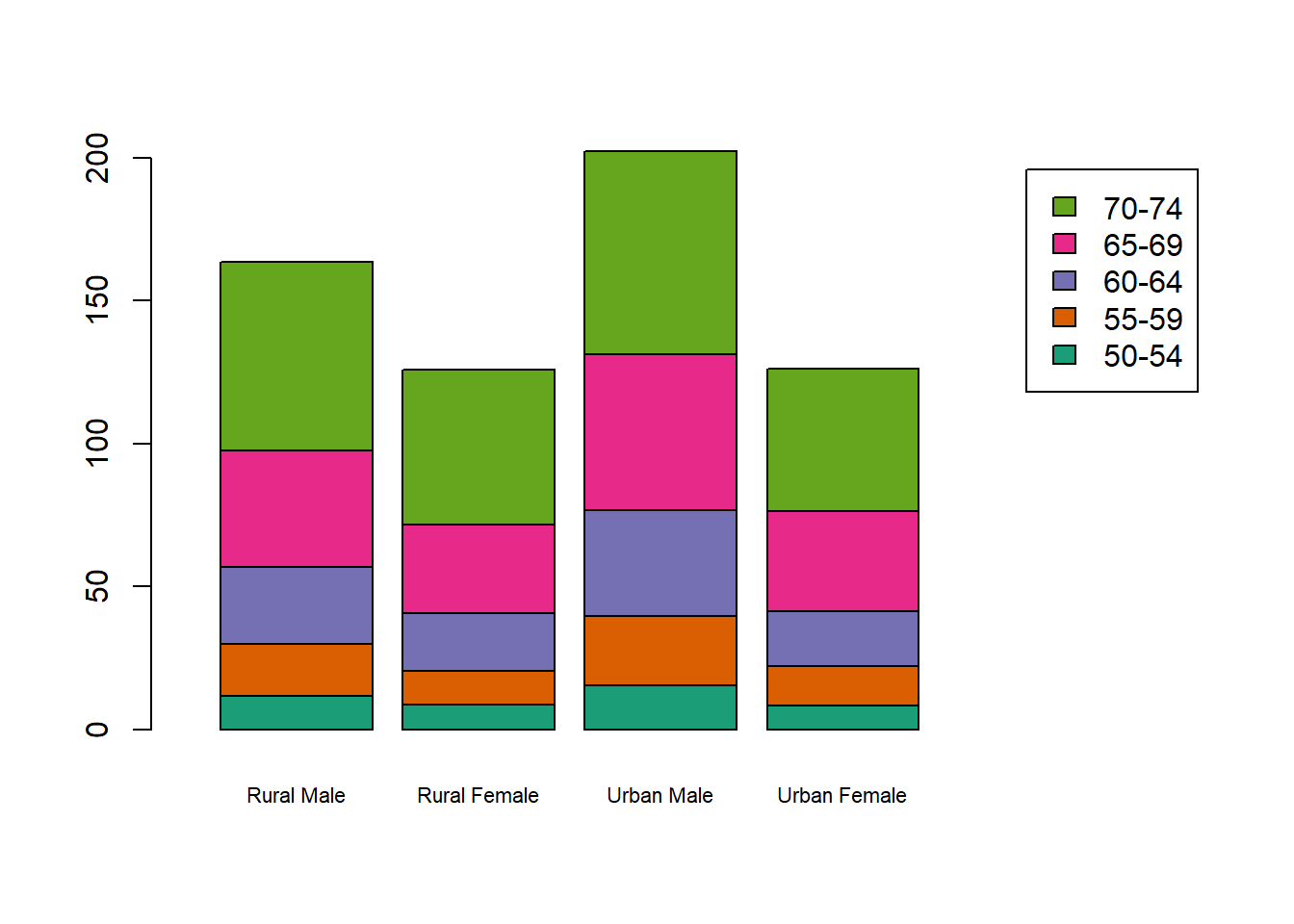
# Barras agrupadas
barplot(VADeaths, col = cl[1:5], border = "black", xlim=c(0,28.5),
cex.names = 0.7, beside = TRUE,
legend = rownames(VADeaths))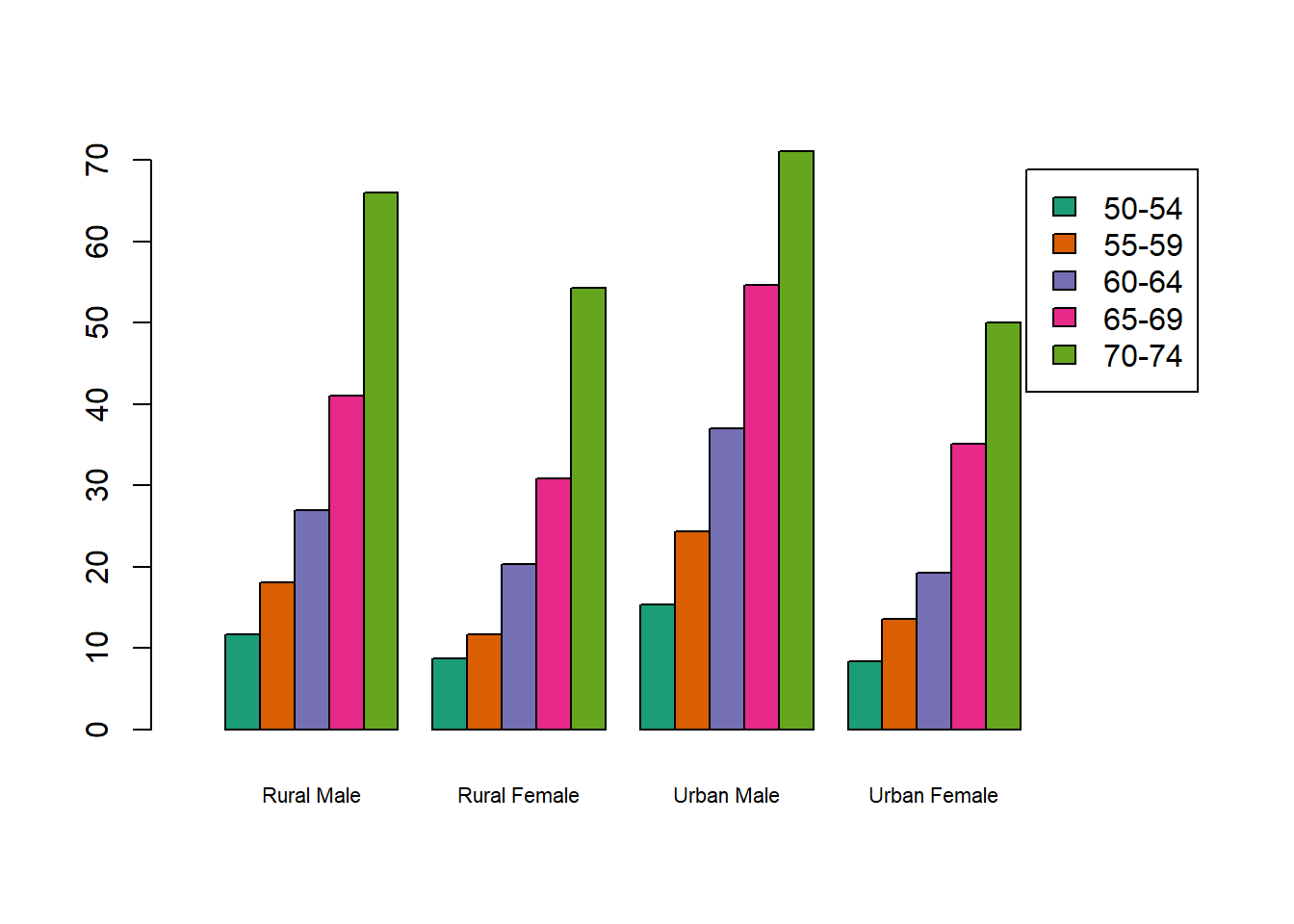
En el caso de tener variable continuas, se necesita agrupar los valores en intervalos usando la función cut, de lo contrario tendremos tantas barras como valores diferentes haya en la variable.
barplot(table(cut(iris$Sepal.Length, breaks = seq(4,8, by=1))),
col = cl[5], border = "black",
main = "Sepal Length Iris Dataset",
xlab="Sepal Length",
ylab="Count")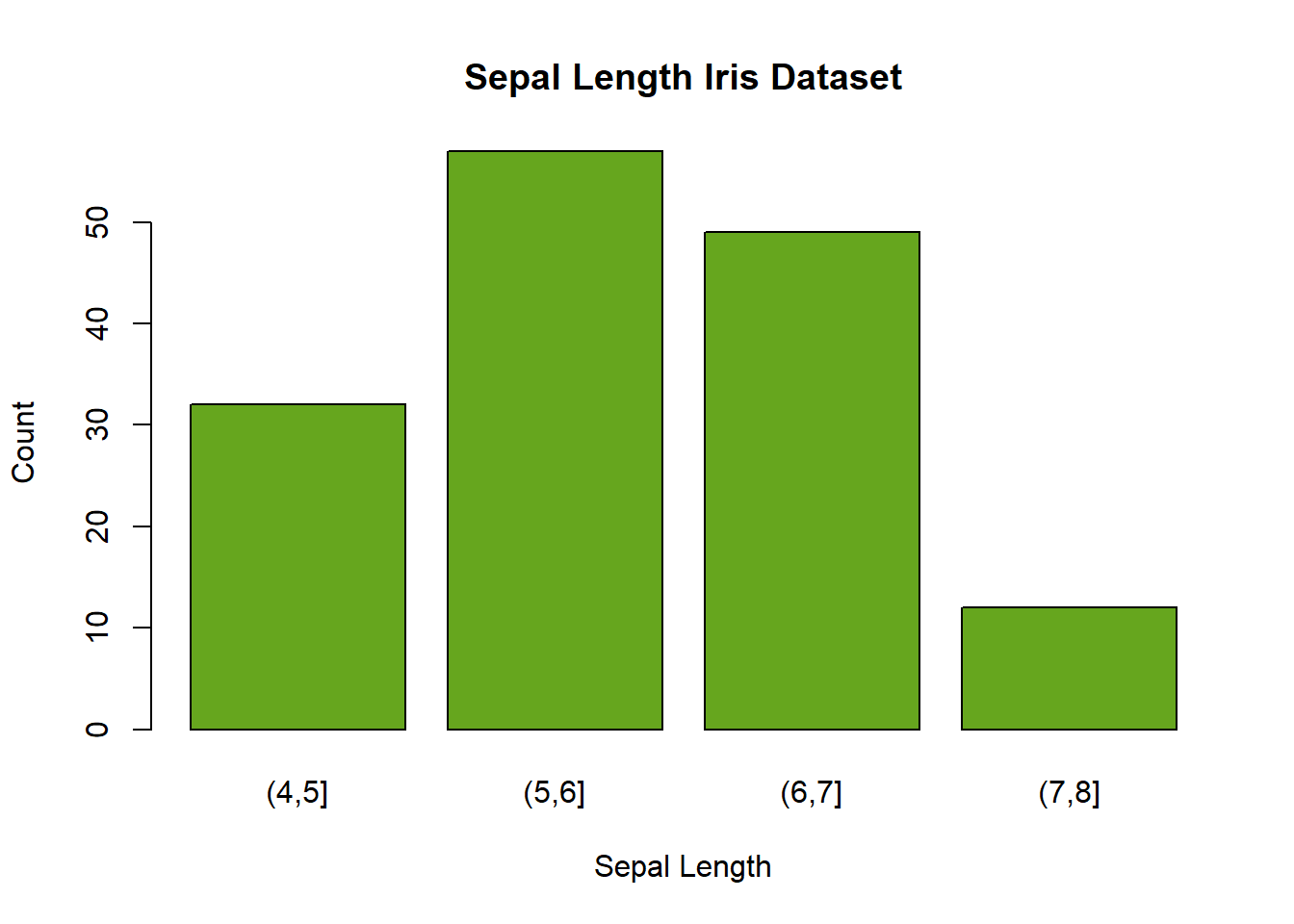
5.1.3 Histograma
Los histogramas son la forma más común de representar vectores numéricos(no necesariamente enteros). Para crear histogramas con R base usamos la función hist(). Los parámetros de la función son los siguientes.
| Argumentos | Descripción |
|---|---|
x |
Vector de valores. |
breaks |
Tamaño de los bin a ser calculados: c(0,n, k). |
freq |
Valor lógico que indica si se van a graficar frecuencias(TRUE) o probabilidades. |
col, border |
Color de llenado de los bins y contornos. |
hist(iris$Sepal.Length, breaks = seq(4,8, by=1),
main = "Histogram from Iris Sepal Length data set",
xlab = "Sepal Length",
col = "blue",
ylab = "Frecuencia")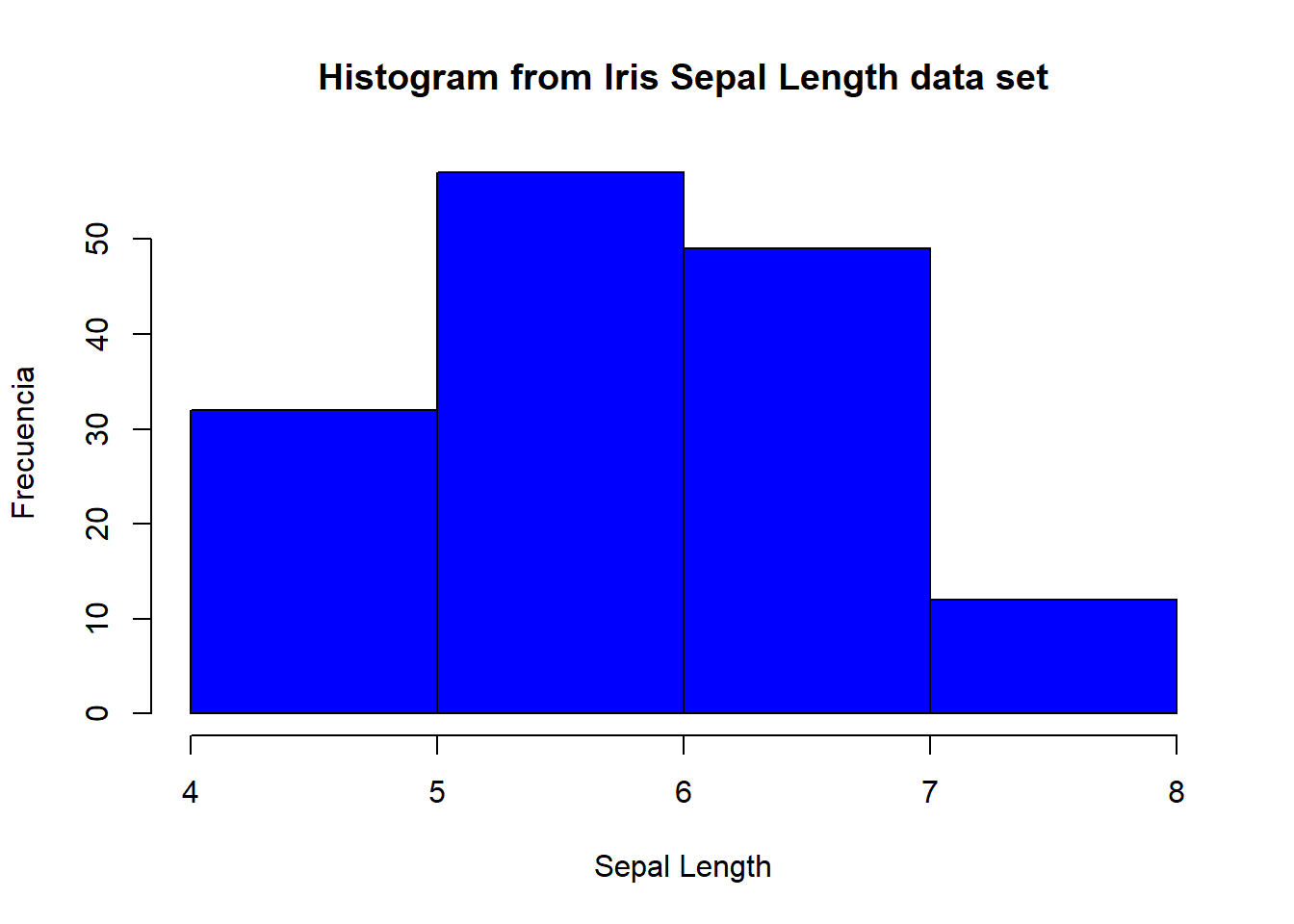
hist(x = ChickWeight$weight,
main = "Histograma peso de Pollos",
xlab = "Peso",
ylab = "Frecuencia",
breaks = 20,
xlim = c(0,500),
col = "khaki1",
border = "orange2")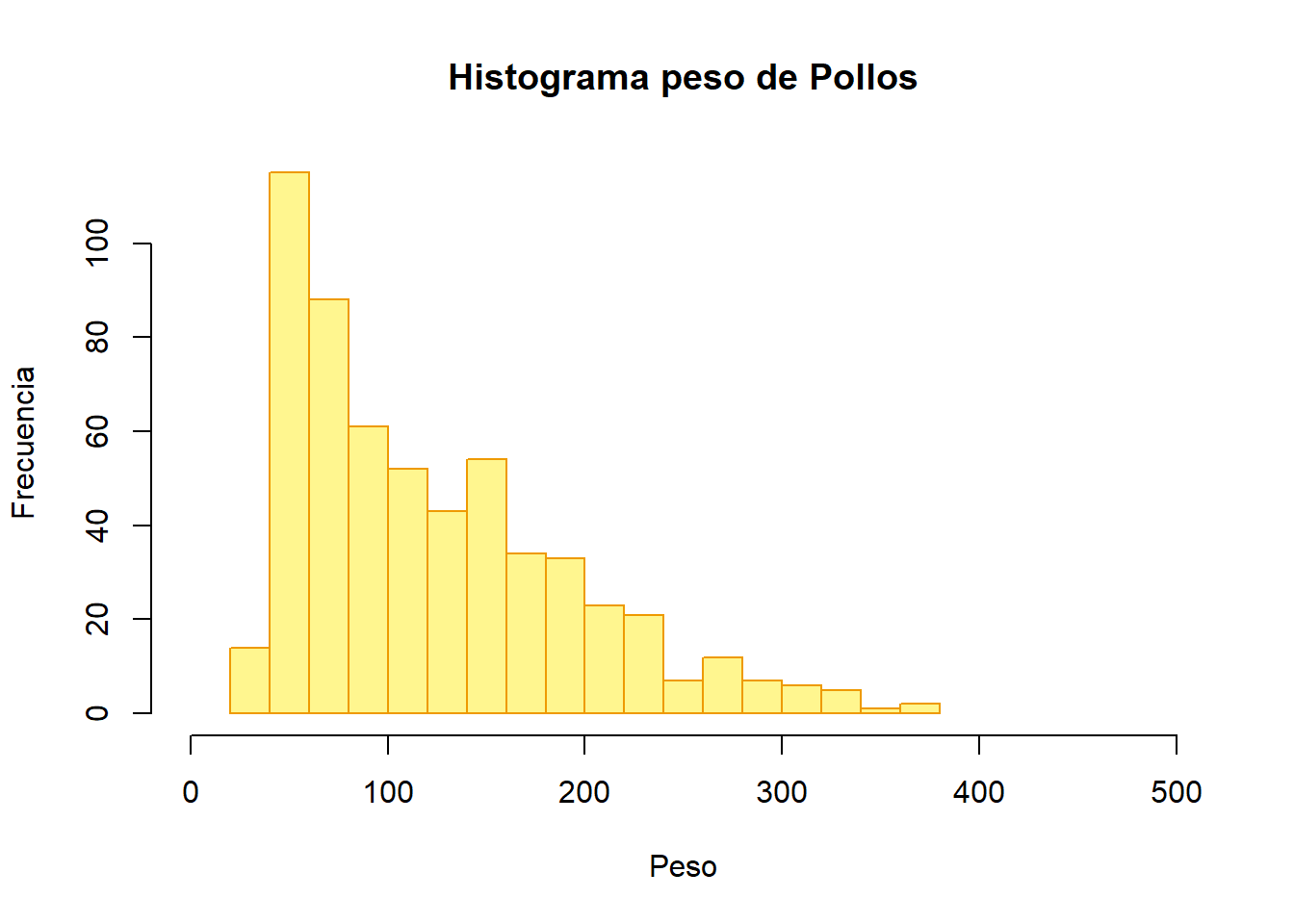
También podemos graficar dos histogramas en uno indicando colores contrastantes.
hist(x = ChickWeight$weight[ChickWeight$Diet==1],
main = "Histograma peso de Pollos",
xlab = "Peso",
ylab = "Frecuencia",
breaks = 20,
xlim = c(0,500),
col = gray(0,0.5))
hist(ChickWeight$weight[ChickWeight$Diet==2],
breaks = 30,
add = TRUE, #para agregarlo al plot anterior
col = gray(1,0.8))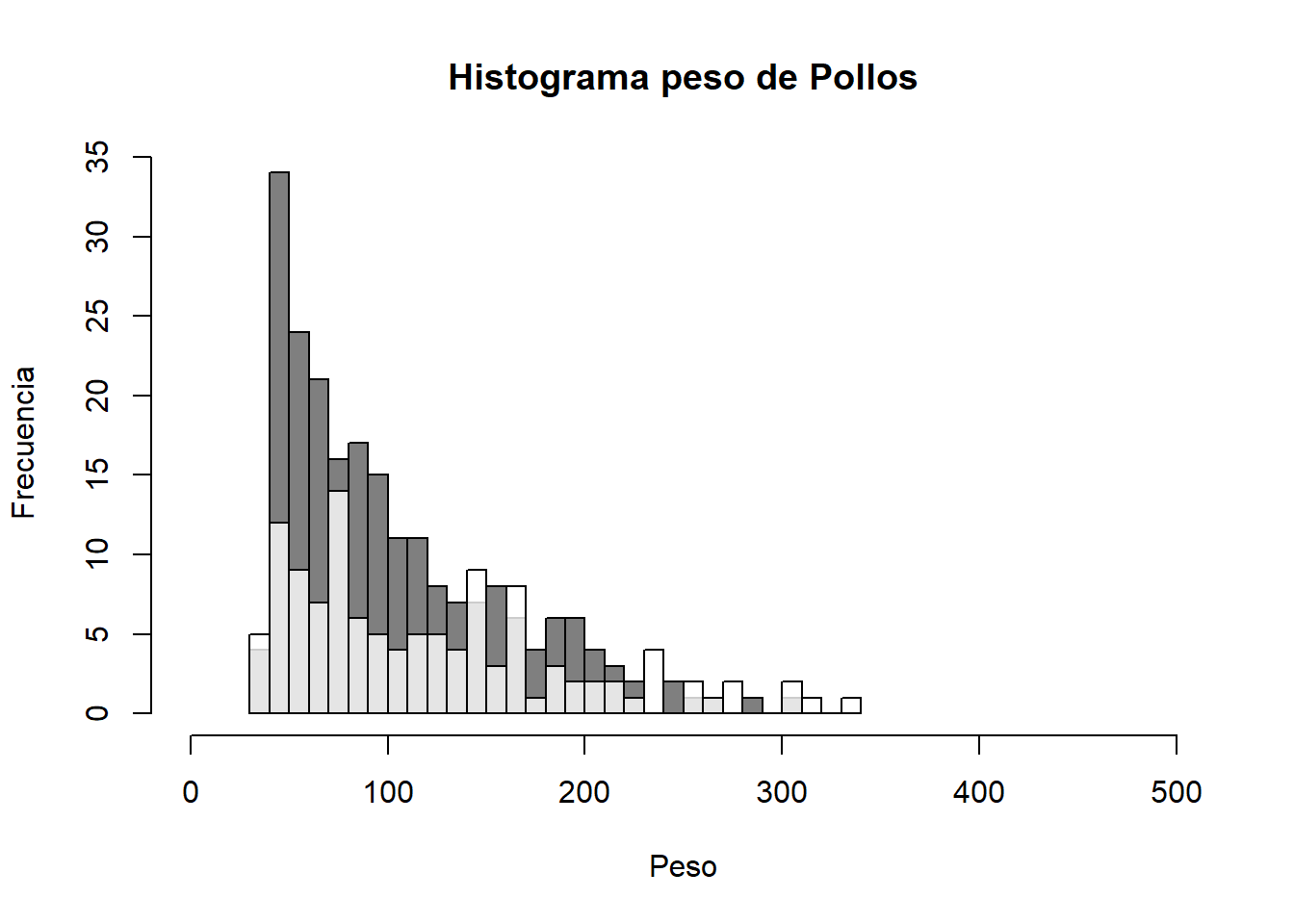
hist(x = ChickWeight$weight[ChickWeight$Diet==1],
main = "Histograma peso de Pollos",
xlab = "Peso",
ylab = "Frecuencia",
breaks = 20,
xlim = c(0,500),
col = "khaki3",
border = "orange2")
hist(ChickWeight$weight[ChickWeight$Diet==2],
breaks = 30,
add = TRUE, #para agregarlo al plot anterior
col = "khaki1")
legend("topright", c("Dieta 1", "Dieta 2"), col=c("khaki3", "khaki1"), lwd=10)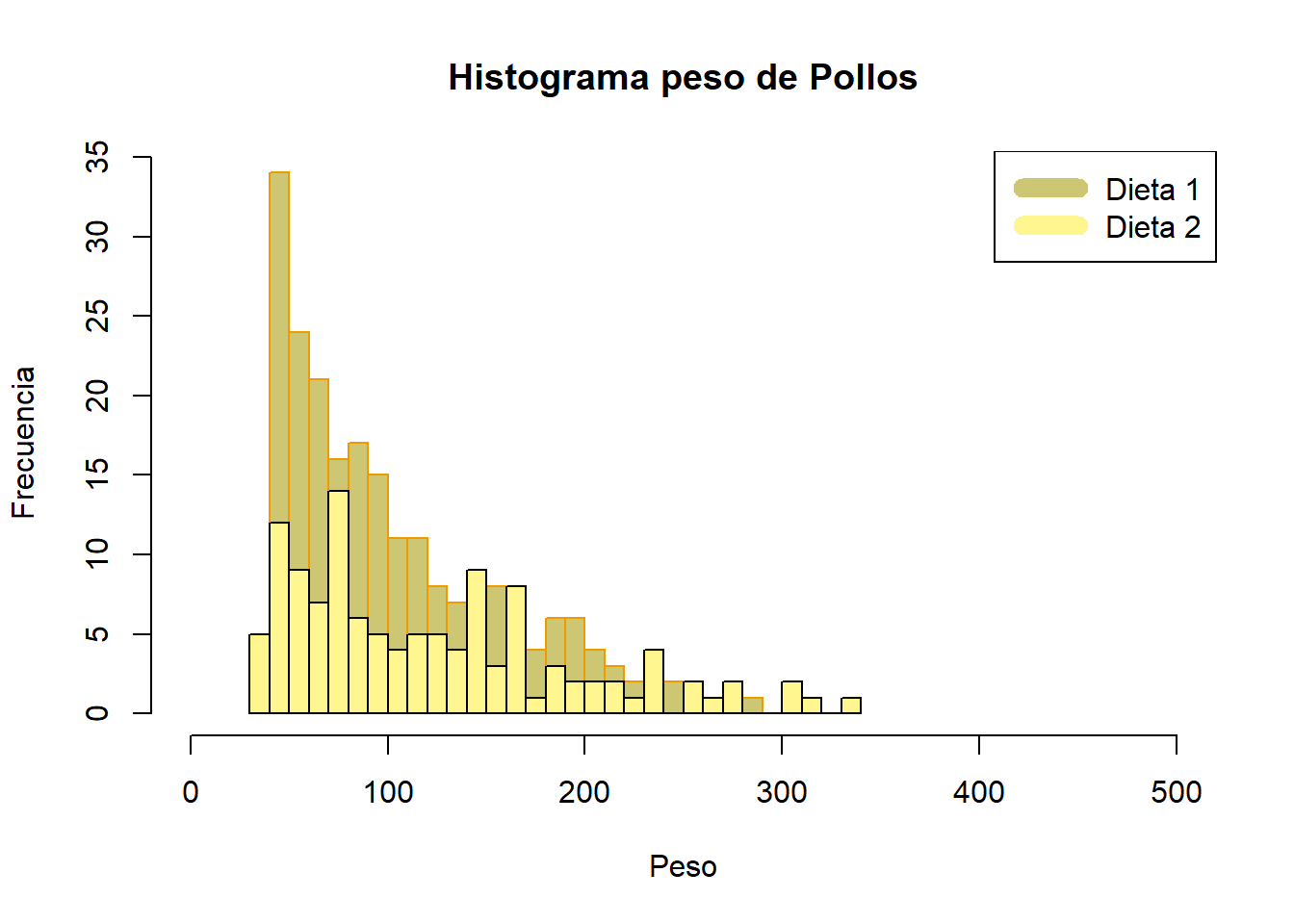
5.1.4 Boxplot
Los boxplot o diagramas de caja y bigotes son útiles para graficar la distribución de los datos represenando los cuantiles. La caja central muestra el rango intercuantil \(IQR\), es decir todos los valores entre el primer y tercer cuantil, marca también la mediana con una línea horizontal. Afuera de la caja se muestran dos bigotes que van del tercer o primer cuantil \(\pm\) 1.5 veces el \(IQR\). Este valor de \(1.5\) es el de default pero se puede modificar. Los puntos que se llegan a marcar fuera de los bigotes son los outliers de los datos. Estos gráficos son útiles para resumir la distribución y la asimetría de los datos alrededor de la media. Cajas muy grandes representan que los datos están más dispersos.
La función en R base para hacer un boxplot es boxplot.
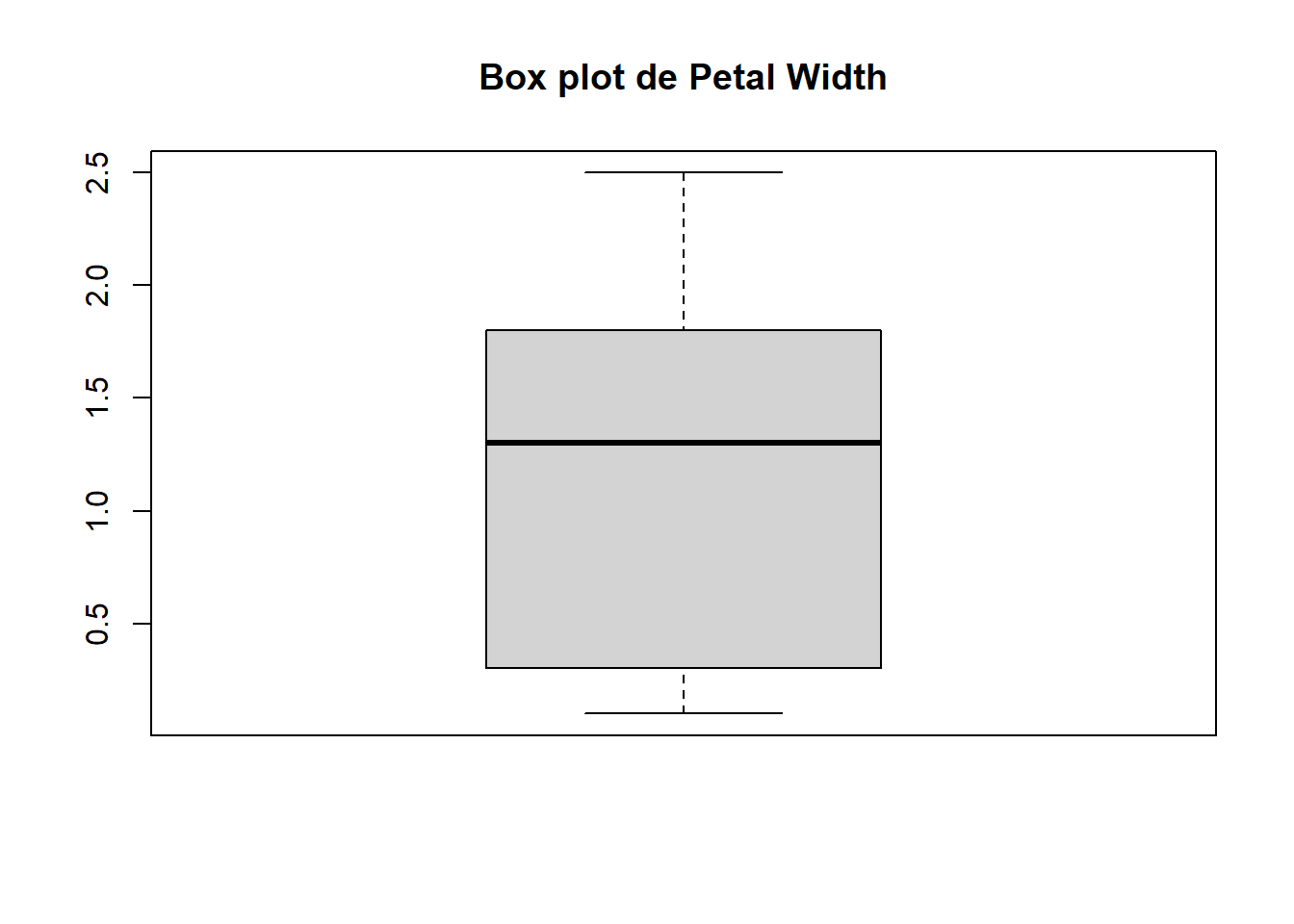
Podemos observar que la mediana del ancho de pétalos es un poco menor que \(1.5\) y que tiene una dispersión muy grande, más del \(50\%\) de los valores están entre el \(0.3\) y \(0.7\). También de esta gráfica podemos ver que los datos están muy sesgados ya que el rango entre el primer cuantil y la mediana es mayor que entre la mediana y el tercer cuantil, la variabilidad del ancho es mayor para valores pequeños.
En estos plots podemos graficar varias cajas a la vez.
boxplot(iris$Sepal.Length ~ iris$Species,
main = "Box plot de la longitud Sepal para cada especie",
xlab = "Especies",
ylab = "Sepal Length")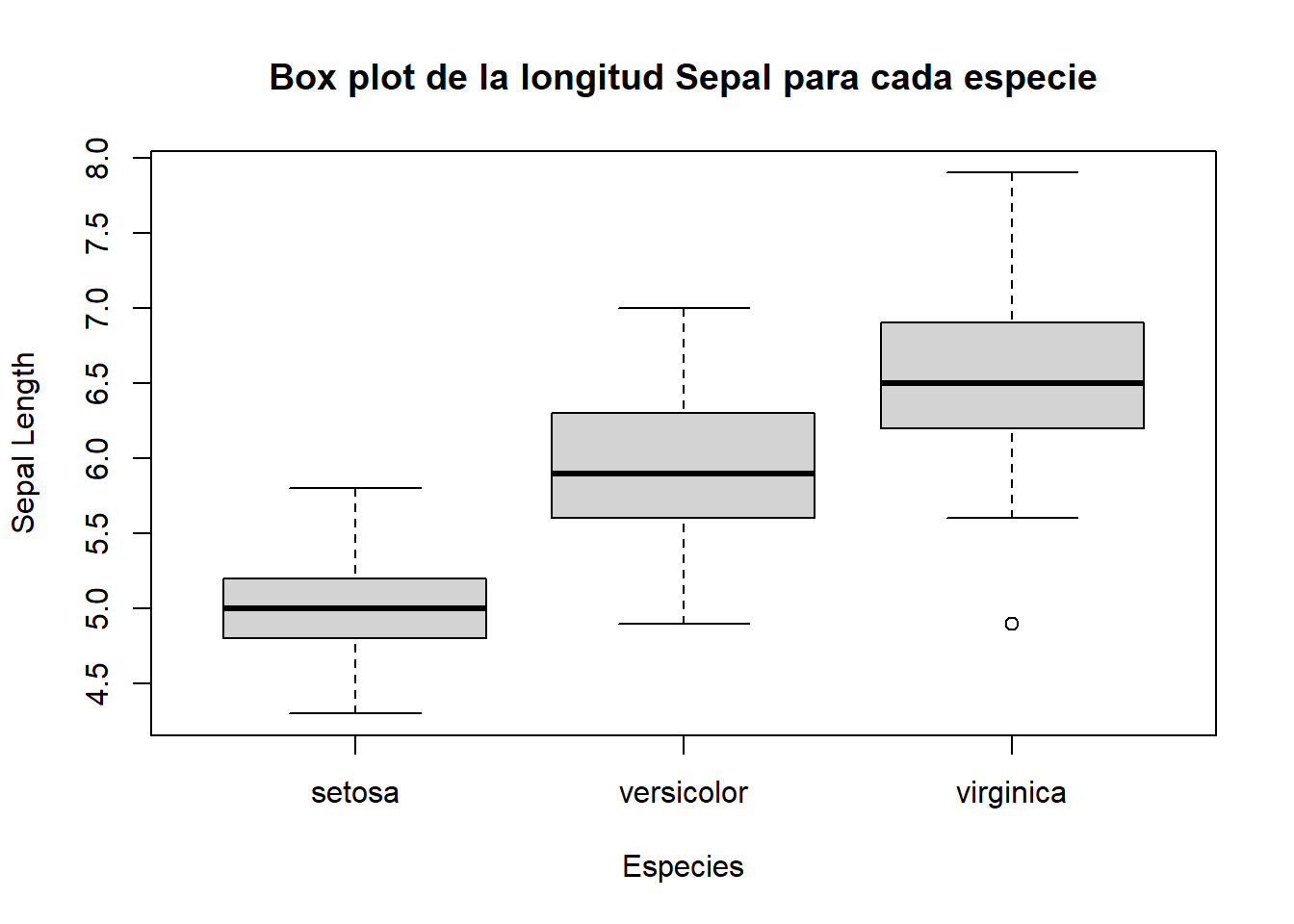
# Boxplots horizontales
boxplot(iris$Sepal.Length ~ iris$Species,
main = "Box plot de la longitud Sepal para cada especie",
xlab = "Especies",
ylab = "Sepal Length",
horizontal = TRUE)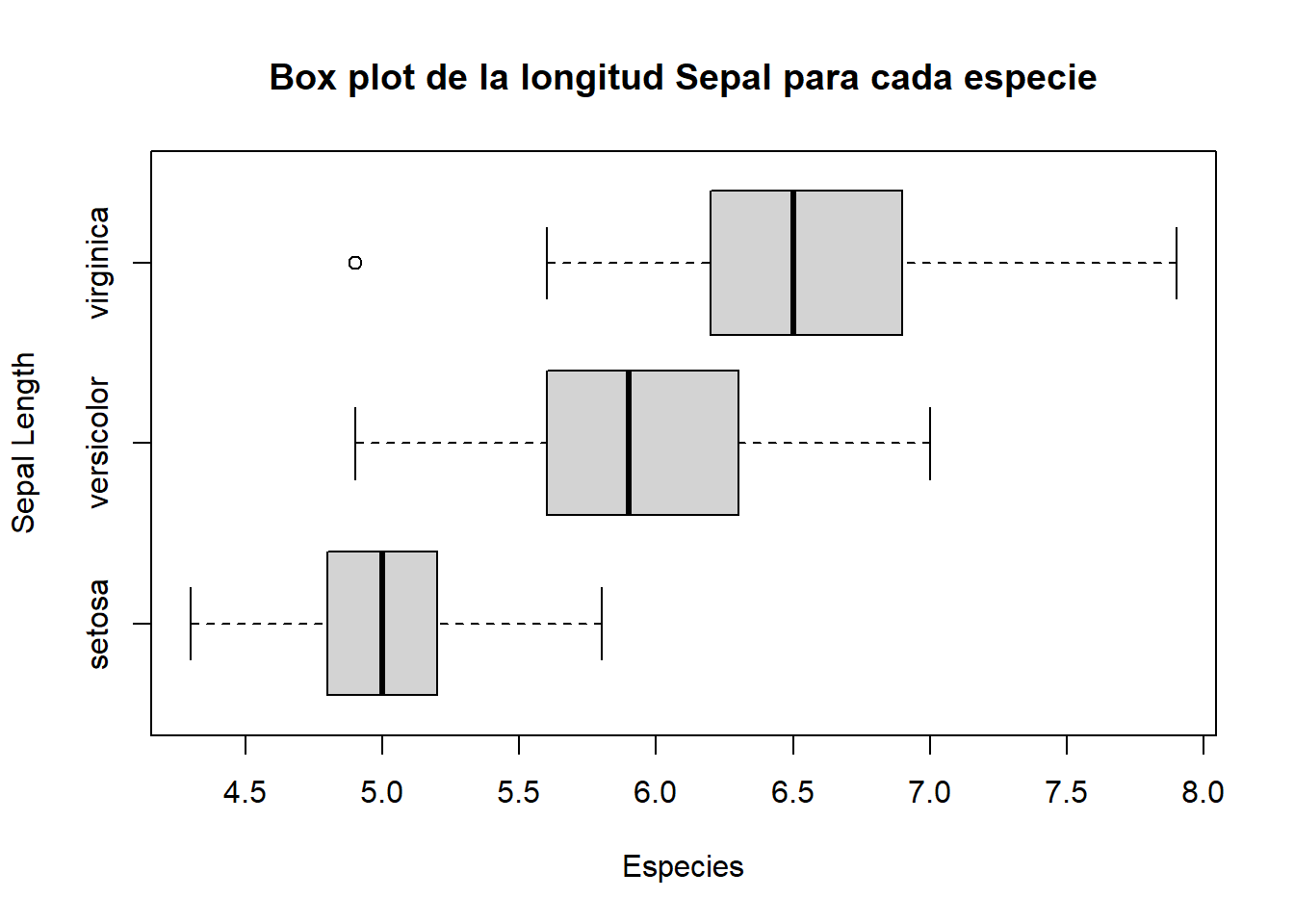
# Remover el marco
boxplot(iris$Sepal.Length ~ iris$Species,
main = "Box plot de la longitud Sepal para cada especie",
xlab = "Especies",
ylab = "Sepal Length",
frame = FALSE)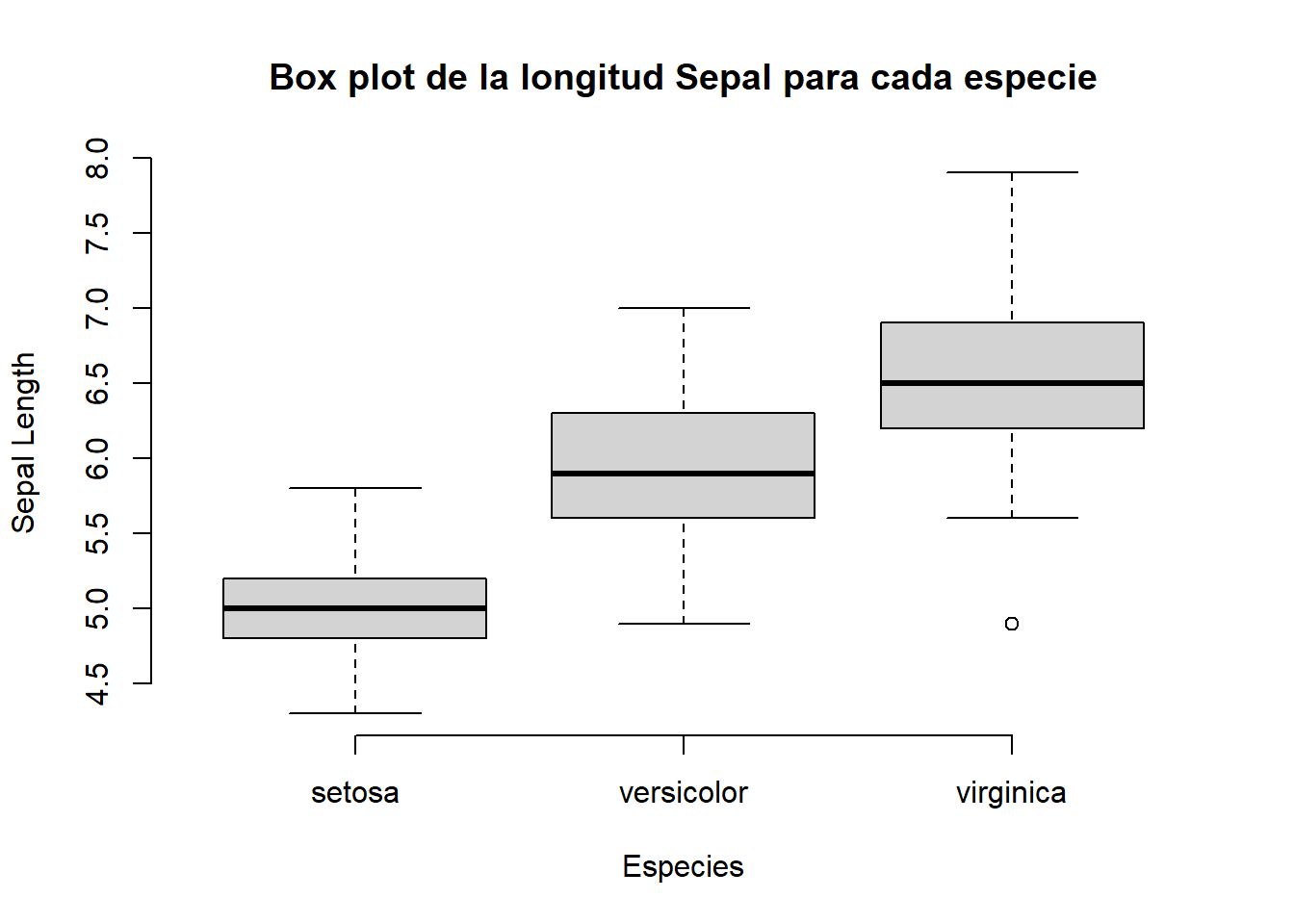
# Notched
boxplot(iris$Sepal.Length ~ iris$Species,
main = "Box plot de la longitud Sepal para cada especie",
xlab = "Especies",
ylab = "Sepal Length",
notch = TRUE)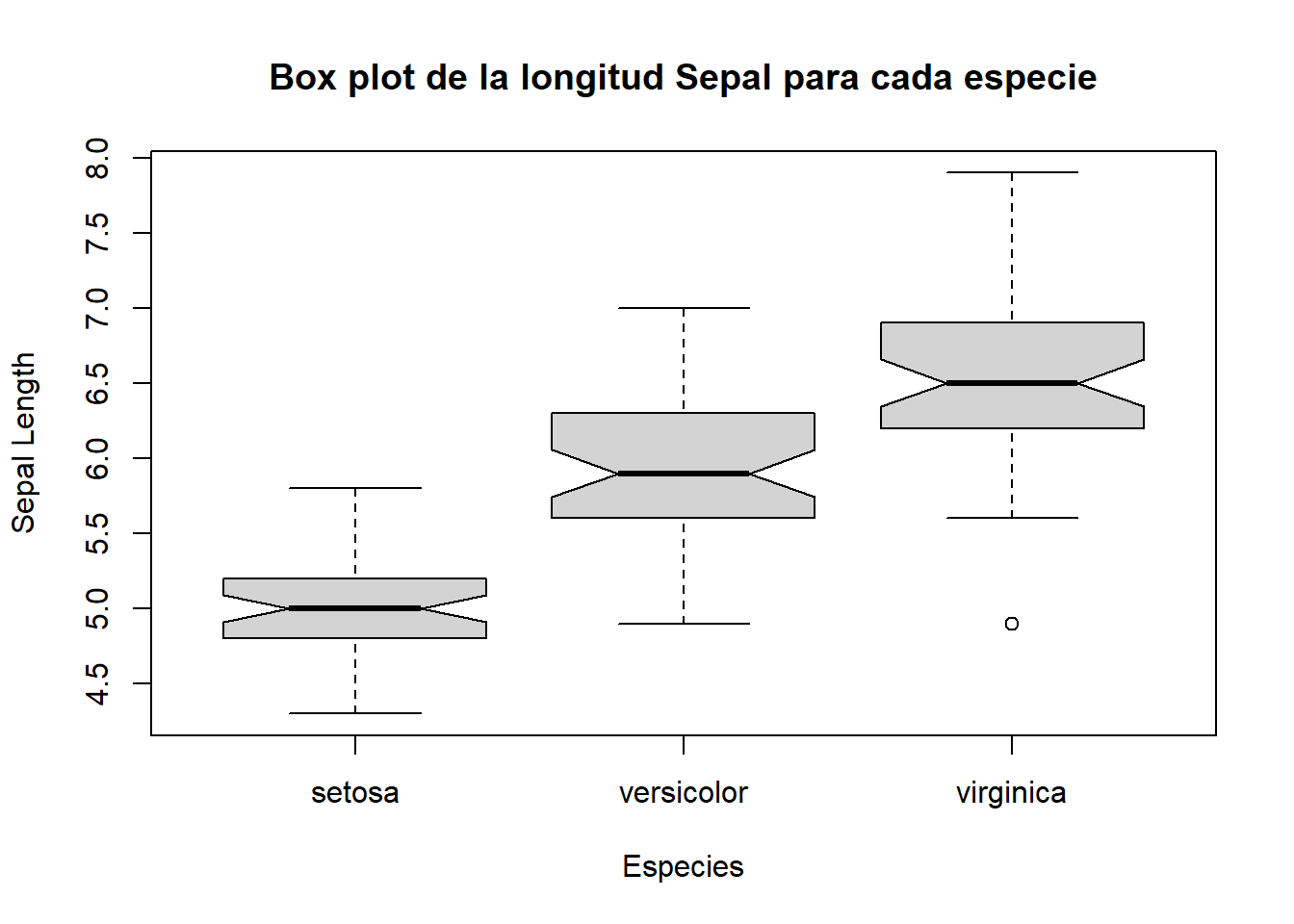
Agregar la opción nocth nos ayuda a tener una idea de si las medianas son diferentes, si no se intersectan las cajas, entonces es evidencia de diferencia entre las medianas.
# Llenar por colores
boxplot(iris$Sepal.Length ~ iris$Species,
main = "Box plot de la longitud Sepal para cada especie",
xlab = "Especies",
ylab = "Sepal Length",
col = c("#FF33CC", "#CC66FF", "#33C9CC"))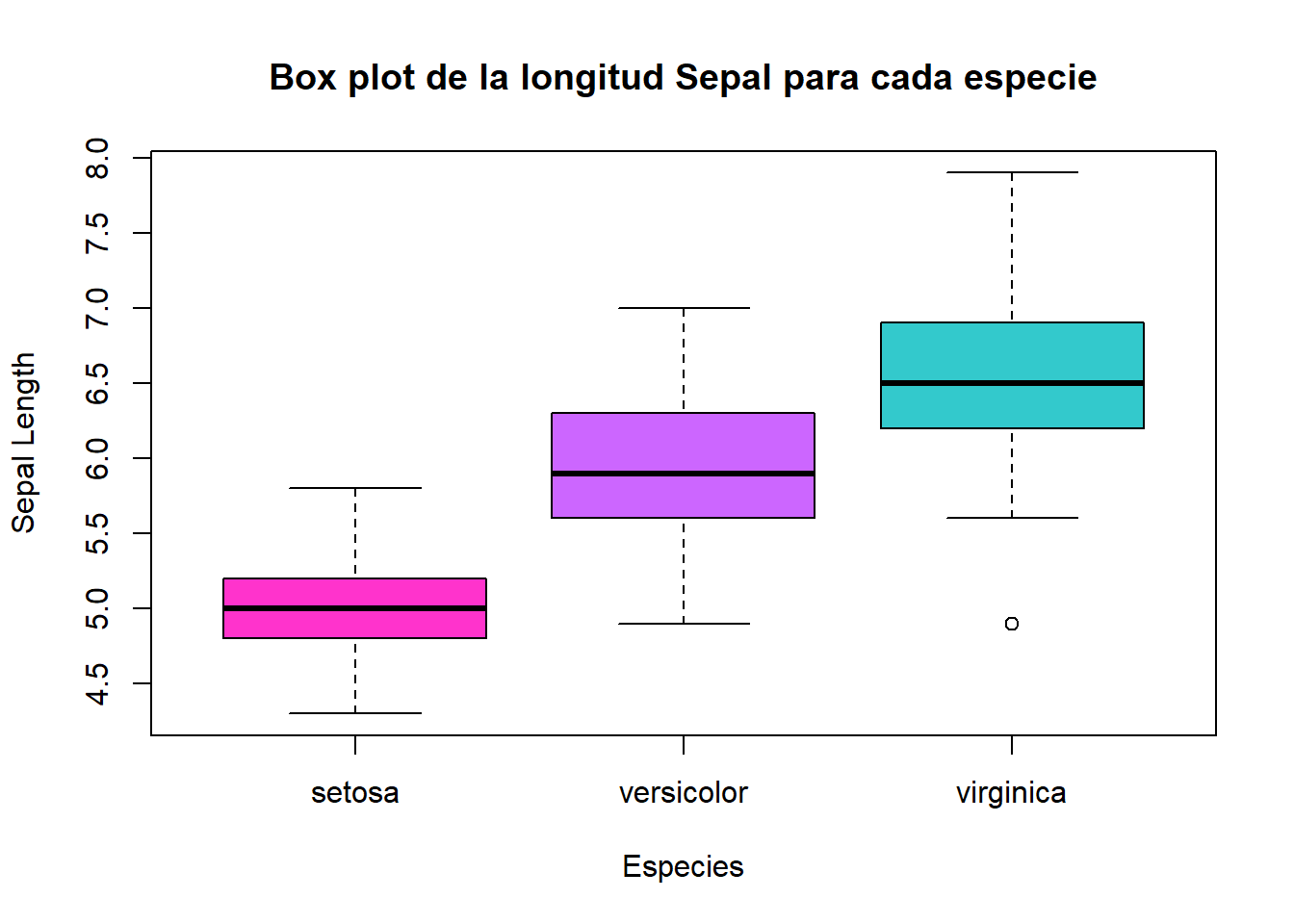
# Contornos
boxplot(iris$Sepal.Length ~ iris$Species,
main = "Box plot de la longitud Sepal para cada especie",
xlab = "Especies",
ylab = "Sepal Length",
border = c("#FF33CC", "#CC66FF", "#33C9CC"),
col = "white")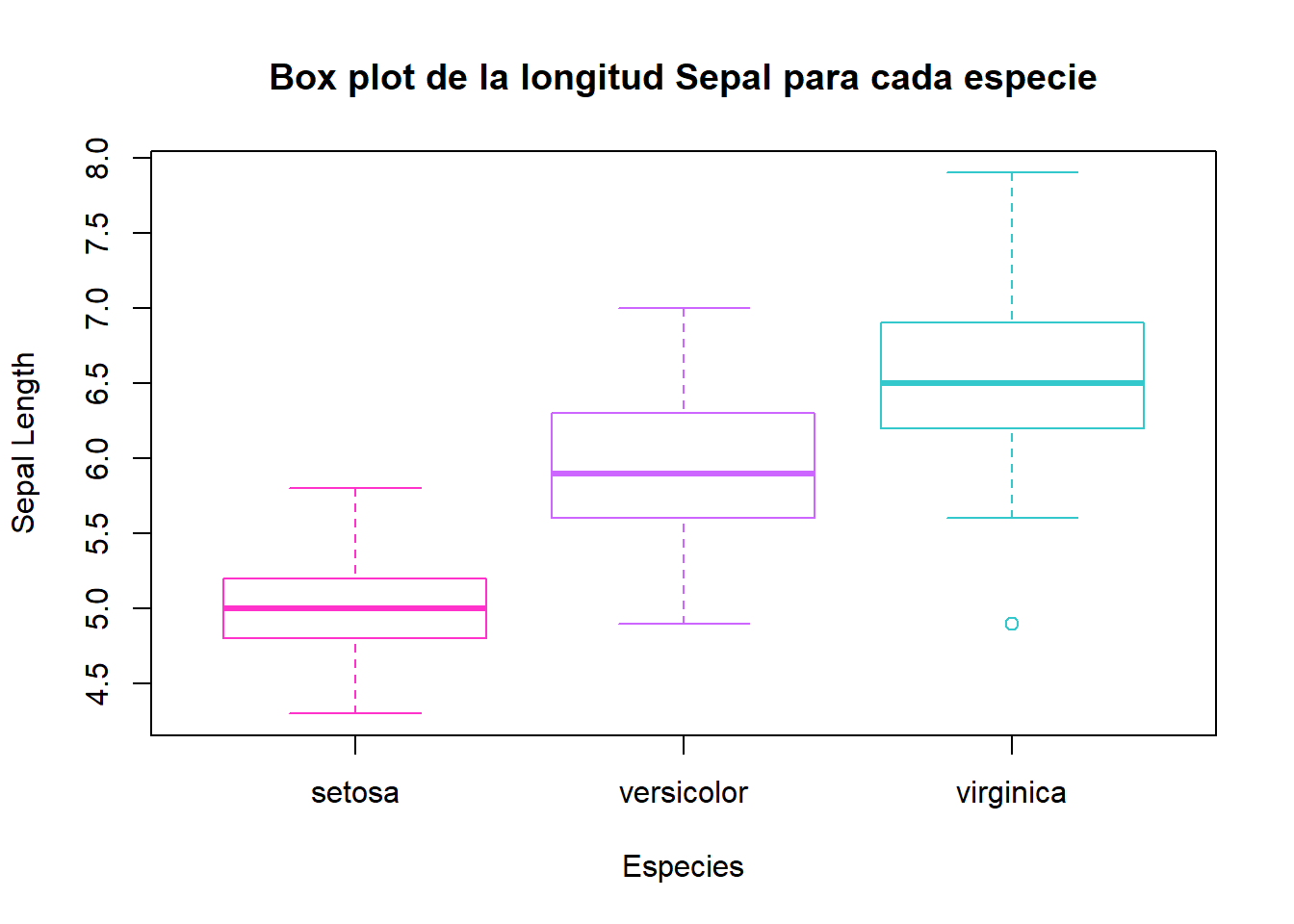
# Leyendas
boxplot(iris$Sepal.Length ~ iris$Species,
main = "Box plot de la longitud Sepal para cada especie",
xlab = "Especies",
ylab = "Sepal Length",
col = c("#FF33CC", "#CC66FF", "#33C9CC")
)
legend("topleft", c("Setosa", "Versicolor", "Virginica"), border="black", fill = c("#FF33CC", "#CC66FF", "#33C9CC"))
5.1.5 Curvas y gráficos de baja dimensión
| Funciones | Acción |
|---|---|
points(x, y) |
Agregar puntos |
abline(), segments() |
Añade líneas o segmentos |
arrows() |
Añade flechas |
curve() |
Añade curvas que representan funciones |
rect(),polygon() |
Añade rectángulos o polígonos |
text(), mtext() |
Añade texto a los plots o fuera de ellos |
legend() |
Agrega leyendas |
axis() |
Agrega ejes |
5.1.5.1 Points()
# Vectores a graficar
height <- c(156, 175, 160, 172, 159, 165, 178)
weight <- c(65, 74, 69, 72, 66, 75, 75)
id <- c("andrew", "heidi", "becki", "madisen", "david", "vincent", "jack")
# Plot vacío
plot(x = 1,
type = "n",
xlim = c(155, 180),
ylim = c(65, 80),
pch = 16,
xlab = "Height",
ylab = "Weight",
main = "Añadir puntos al plot con points()")
points(x = height, y = weight,
pch = 16,
col = "blue")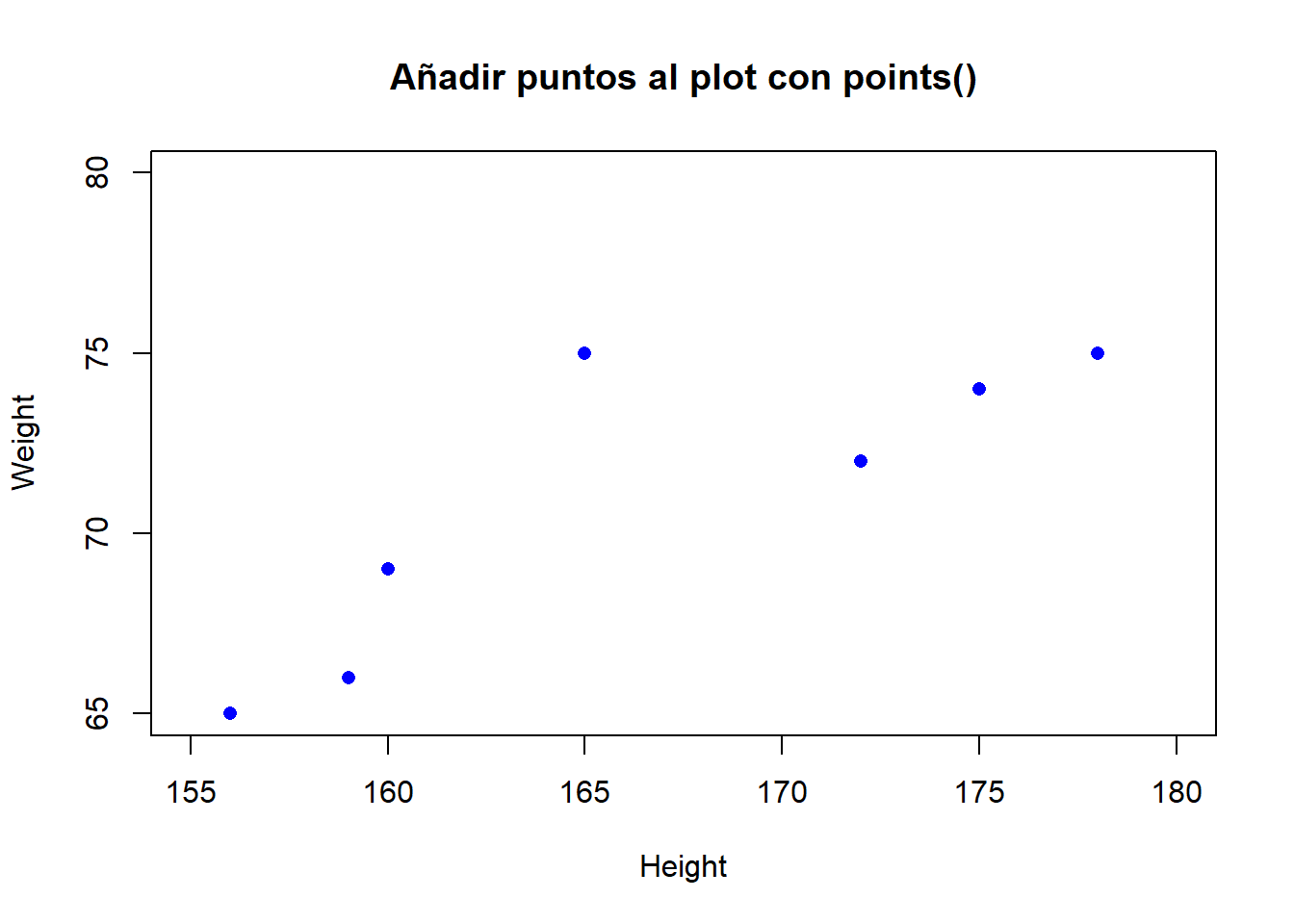 ####
#### text()
# Vectores
height <- c(156, 175, 160, 172, 159, 165, 178)
weight <- c(65, 74, 69, 72, 66, 75, 75)
id <- c("andrew", "heidi", "becki", "madisen", "david", "vincent", "jack")
# Plot
plot(x = height,
y = weight,
xlim = c(155, 180),
ylim = c(65, 80),
pch = 16,
col = "red")
# Agregar etiquetas
text(x = height,
y = weight,
labels = id,
pos = 3)  ####
#### abline()
# Crear plot con la base de datos de Chickens
plot(x = ChickWeight$Time,
y = ChickWeight$weight,
col = gray(.3, .5),
pch = 16,
main = "Combinar texto con números con la función paste()",
xlab = "Tiempo",
ylab = "Pesos")
# Agregar líneas de referencia
abline(h = mean(ChickWeight$weight),
lty = 2)
# Agregar texto
text(x = 3,
y = mean(ChickWeight$weight),
labels = paste("Media de peso =",
round(mean(ChickWeight$weight), 2)),
pos = 3)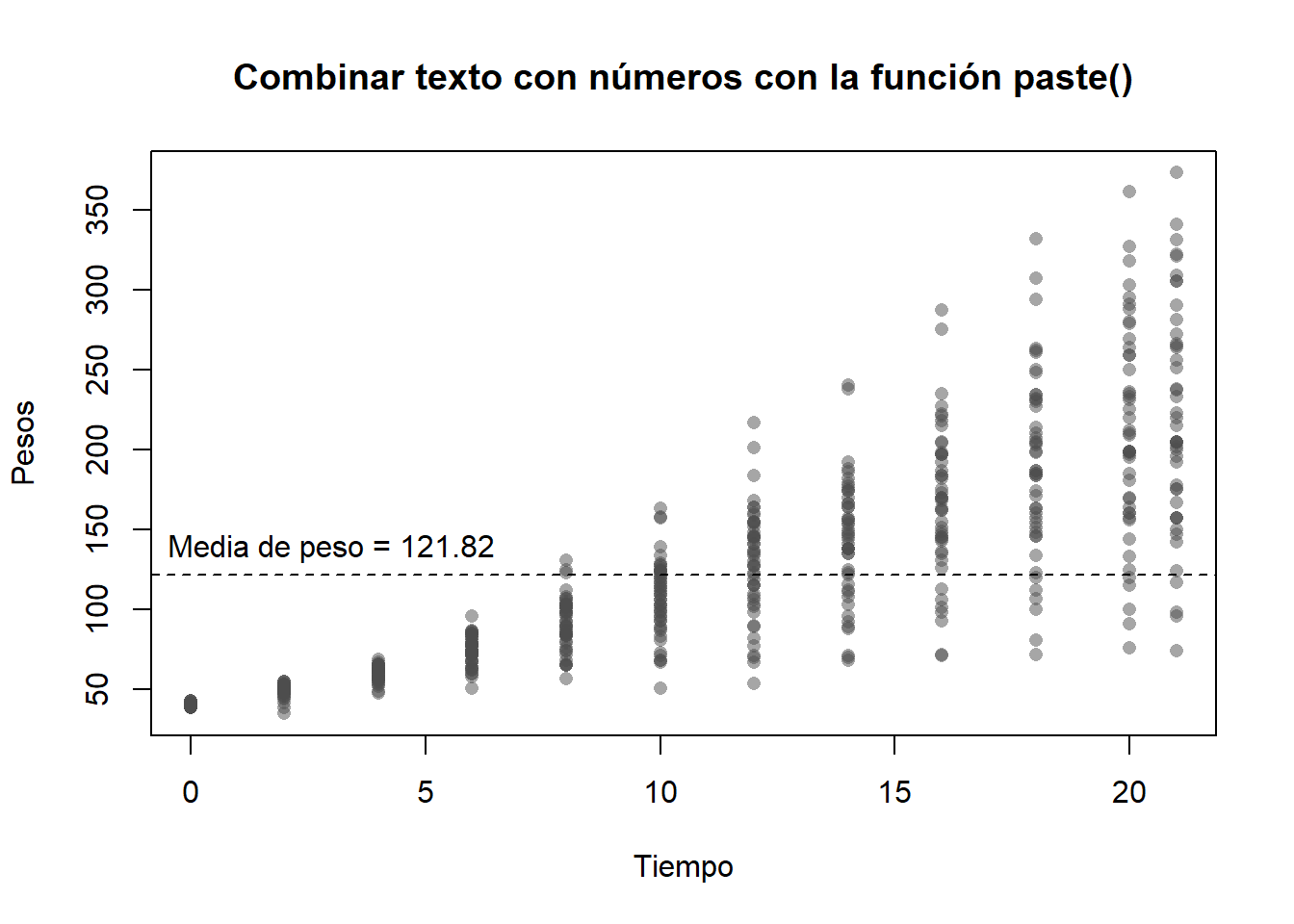 ####
#### curve()
| Argumentos | Acción |
|---|---|
expr |
Nombre de la función escrita como función de la variable x que regresa un vector. Se pueden usar expresiones como expr = $x^2$, expr = x + 7, o programar funciones con expr = mi.funcion, donde my.funcion es una función que se definió previamente (ej; mi.funcion <- function(x) {dnorm(x, mean = 10, sd = 3)) |
from, to |
Valores iniciales (from) y finales (to) de la variable \(x\) a ser graficados. |
add |
Valor lógico que indica cuando se añade una curva o no al plot existente. Si es add = FALSE, entonces la función curve() creará un nuevo plot. Si add = TRUE, entonces curve() agregará la curva al plot existente. |
lty, lwd, col |
Argumentos adicionales como tipo de línea, grosor, color. |
funcion1 <- function(x){x^2-1}
funcion2 <- function(x){-x^2+100}
curve(funcion1, from = -10, to = 10, lwd = 2, lty = 1, col = "red")
curve(funcion2, add = TRUE, lwd = 2, lty = 1, col = "blue")
legend("topright", c("Función 1: $x^2+1$", "Función 2: $-x^2+100$"), col = c("red", "blue"), lwd = 3)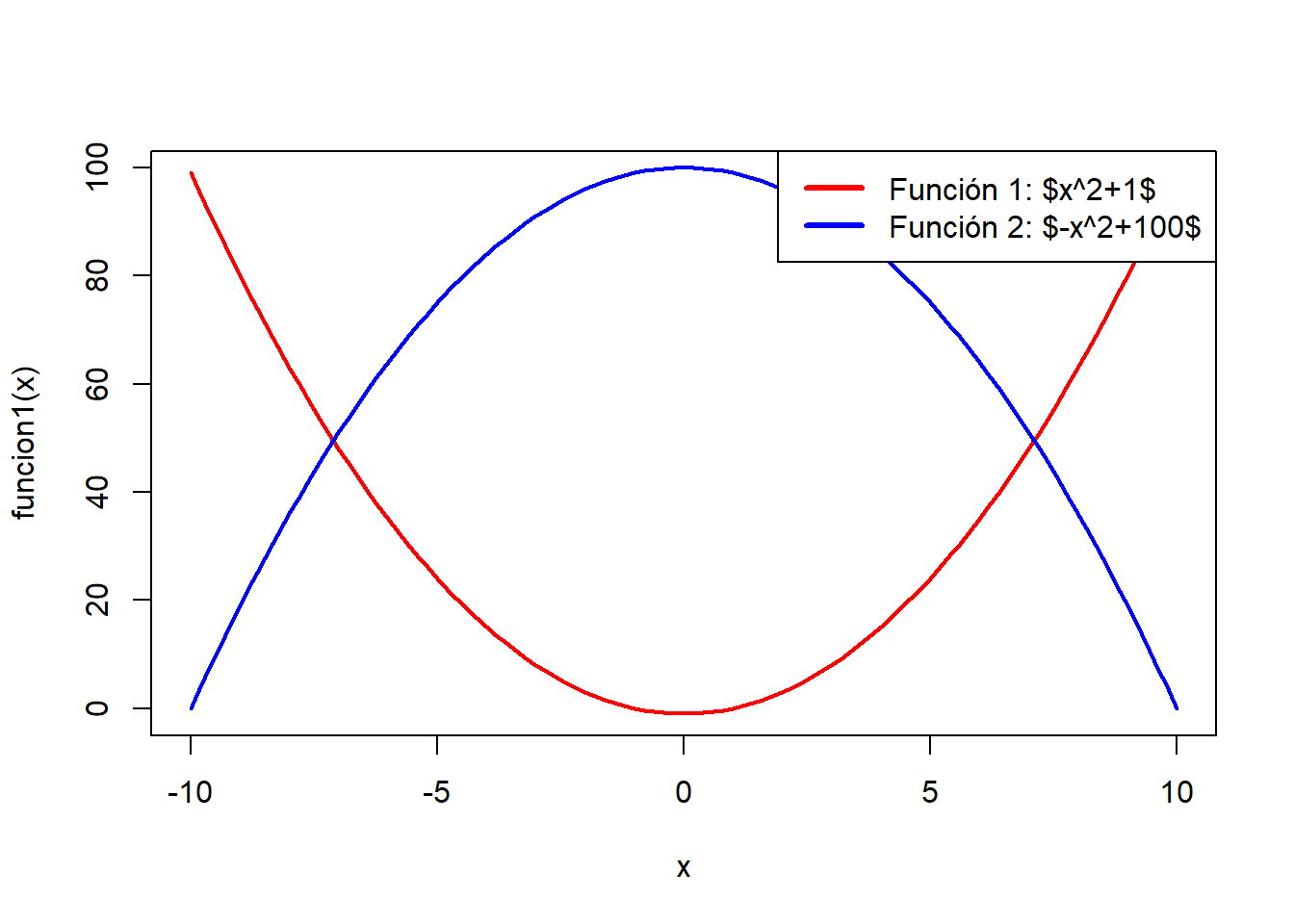
# Crear plot vacío
plot(1,
xlim = c(-5, 5), ylim = c(-5, 5),
type = "n",
main = "Plotting function lines with curve()",
ylab = "", xlab = "")
#Agregar ejes x y y
abline(h = 0)
abline(v = 0)
# Configurar colores
cl <- c("#FF33CC", "#CC66FF", "#33C9CC")
# x ^ 2
curve(expr = x^2, from = -5, to = 5,
add = TRUE, lwd = 3, col = cl[1])
# cos(x)
curve(expr = cos, from = -5, to = 5,
add = TRUE, lwd = 3, col = cl[2])
# dnorm(mean = 2, sd = .2)
mi.funcion <- function(x) {return(dnorm(x, mean = 2, sd = .2))}
curve(expr = mi.funcion,
from = -5, to = 5,
add = TRUE,
lwd = 3, col = cl[3])
# Agregar leyendas
legend("bottomright",
legend = c("x^2", "cos(x)", "dnorm(x, 2, .2)"),
col = cl[1:3],
lwd = 3)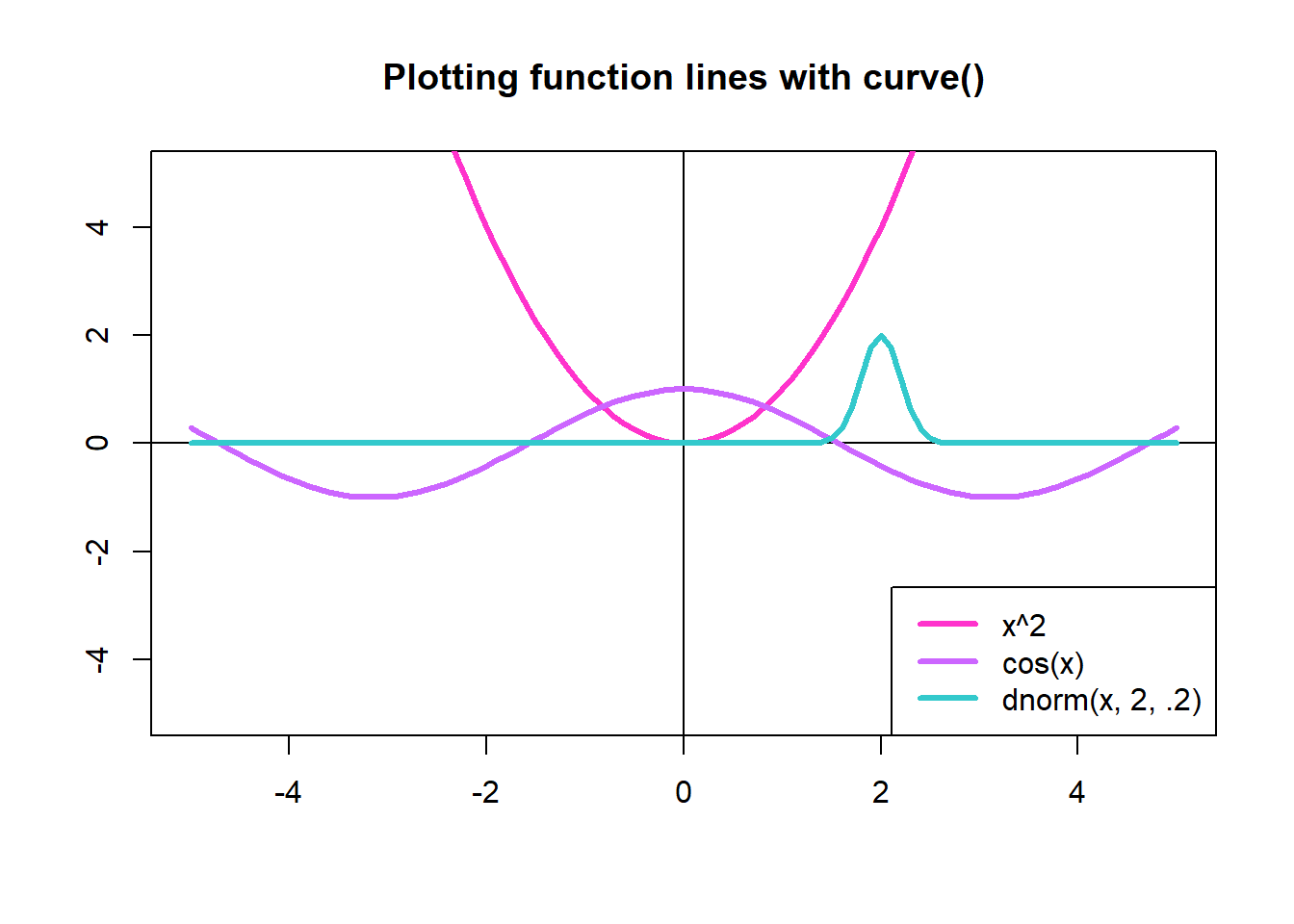
5.1.5.2 Otras funciones
plot(1, xlim = c(1, 100), ylim = c(1, 100),
type = "n", xaxt = "n", yaxt = "n",
ylab = "", xlab = "", main = "Agregar otras figuras a un plot")
text(25, 95, labels = "rect()")
rect(xleft = 10, ybottom = 70,
xright = 40, ytop = 90, lwd = 2, col = "coral")
text(25, 60, labels = "polygon()")
polygon(x = runif(6, 15, 35),
y = runif(6, 40, 55),
col = "skyblue")
text(25, 30, labels = "segments()")
segments(x0 = runif(5, 10, 40),
y0 = runif(5, 5, 25),
x1 = runif(5, 10, 40),
y1 = runif(5, 5, 25),
lwd = 2)
text(75, 95, labels = "symbols(circles)")
symbols(x = runif(3, 60, 90),
y = runif(3, 60, 70),
circles = c(1, .1, .3),
add = TRUE, bg = gray(.5, .1))
text(75, 30, labels = "arrows()")
arrows(x0 = runif(3, 60, 90),
y0 = runif(3, 10, 25),
x1 = runif(3, 60, 90),
y1 = runif(3, 10, 25),
length = .1, lwd = 2)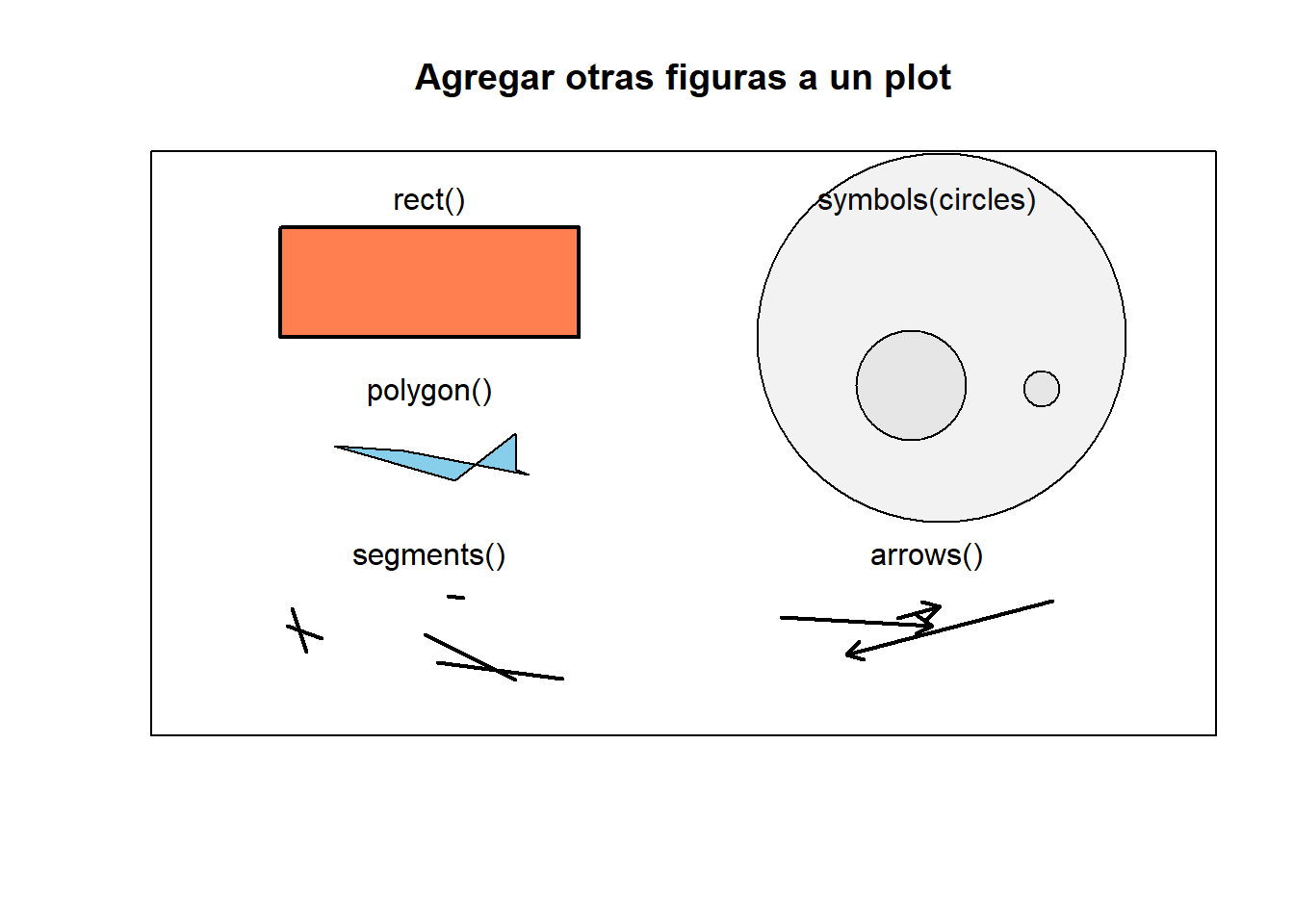
5.1.6 Guardar plots
En el espacio a la derecha abajo, existe la opción de exportar los plots pero también se pueden guardar y configurar desde la consola o script. Las funciones que se usan son pdf(), png(), jpeg().
| Argumentos | Acción |
|---|---|
file |
La dirección y nombre del archivo a guardar, por ejemplo file = "/Users/haydee/Desktop/plot.pdf". La extensión pdf indica que se guardará como pdf. |
width, height |
El ancho y alto de la imagen en pulgadas. |
dev.off() |
No es un argumento para las funciones, se ejecuta este código al final de crear el plot para terminar de guardar el archivo. |
# Configuración del archivo donde se guardará el plot
pdf(file = "~/R_sites/Seminario_Estadistica/img/funciones.pdf",
width = 10, height = 8)
# Crear plot vacío
plot(1,
xlim = c(-5, 5), ylim = c(-5, 5),
type = "n",
main = "Plotting function lines with curve()",
ylab = "", xlab = "")
#Agregar ejes x y y
abline(h = 0)
abline(v = 0)
# Configurar colores
cl <- c("#FF33CC", "#CC66FF", "#33C9CC")
# x ^ 2
curve(expr = x^2, from = -5, to = 5,
add = TRUE, lwd = 3, col = cl[1])
# cos(x)
curve(expr = cos, from = -5, to = 5,
add = TRUE, lwd = 3, col = cl[2])
# dnorm(mean = 2, sd = .2)
mi.funcion <- function(x) {return(dnorm(x, mean = 2, sd = .2))}
curve(expr = mi.funcion,
from = -5, to = 5,
add = TRUE,
lwd = 3, col = cl[3])
# Agregar leyendas
legend("bottomright",
legend = c("x^2", "cos(x)", "dnorm(x, 2, .2)"),
col = cl[1:3],
lwd = 3)
dev.off()5.1.7 Ejercicios
- Carga la base de datos
rockque contiene las medidas de 48 muestras de rocas. Las variables de esta base de datos son area de los porosarea, perímetro total de los porosperi, formashape, permiabilidadperm. Construye plots que te ayuden a responder las siguientes preguntas.
- Analiza los scatterplots de las variables. ¿Qué relaciones encuentras?
- Contruye scatterplots de los siguientes pares de variables:
areavsperi,perivsshape,shapevsperm. Todos los plots parecen tener dos grupos de individuos. Revisa si todos los grupos tienen los mismos individuos. ¿Cuáles son tus conclusiones? - Usa histogramas para obtener una evidencia clara de la presencia de dos grupos distintos en la población.
- Repite los histogramas usando en esta ocasión las frecuencias relativas (porcentaje) y conecta la parte superior de las barras con líneas.
- Carga la base de datos
airquality. Grafica los niveles de ozono (Ozone) vs temperatura (Temp) con las siguientes instrucciones:
- Los tamaños de las burbujas/puntos son un tercio del nivel de viento para cada observación.
- El color de los marcadores es diferente para cada mes.
- Los ejes van de -5 a 170 horizontalmete y de 50 a 100 verticalmente.
- Dale formato a los ejes y título del plot.
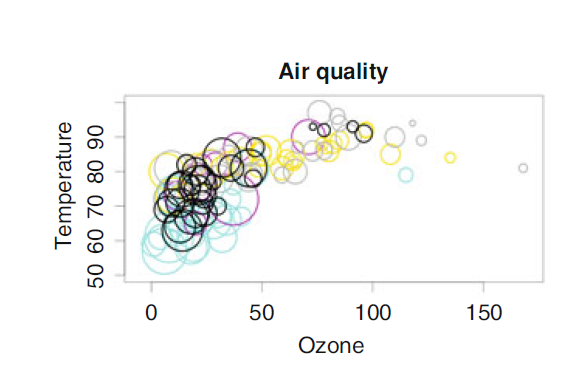
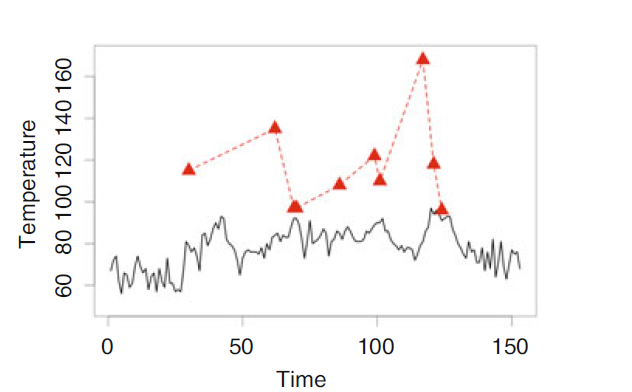
- Con la misma base de datos
airqualitygrafica la evolución de la temperaturaTempcon las siguientes especificaciones:
- La temperatura se representa como una línea sobre el tiempo.
- El tiempo va de la primera observación a la ultima (153).
- Dibuja triángulos solo para aquellos días en que el valor del ozono es más grande que la medida de la temperatura para ese día.
- Añade una leyenda que indique que representa cada línea.
- Da formato a los ejes y título del plot.
- Usando las bases de datos de superhéroes del capítulo anterior:
- Crea un gráfico de barros relativo y absoluto de la cantidad de superhéroes por compañía.
- Crea un gráfico de barros relativo y absoluto de la cantidad de superhéroes hombres y mujeres por compañía.
- Crea un histograma con la frecuencia de la raza de los superhéroes.
- Crea un gráfico con la cantidad de superhéroes buenos, malos y NA. Si solo consideras los personajes femeninos, ¿cómo realizarías un gráfico de pastel con esta información?
- Representa en un gráfico los poderes de los personajes. ¿Qué tipo de gráfico se puede usar para representar esta información?
- Hint: Usa la función
t(as.matrix(base_de_datos)para poder realizar los gráficos. Debes de tener una base de datos adecuada para poder usar las funciones de esta sección.
5.2 ggplot2
El paquete ggplot ayuda a crear gráficas con una mejor calidad. La instrucción es la misma en todos los casos y se puede configurar el tipo de gráfico a realizar. El paquete ggplot2 se basa en la “Gramática de los Gráficos” de Wilkinson. La idea de esto es que cada componente de una gráfica se puede ver como capas independientes, lo que nos permite realizar gráficas paso a paso de manera flexible.

Para hacer un gráfico, basta con proporcionar los argumentos de data y mapping. La instrucción básica para crear un plot es como sigue:
Algunos de los argumentos más comunes de los plots en ggplot son los siguientes:
- Datos:
- Los datos deben de estar en un formato adecuado.
- Cada observación debe estar en una fila diferente.
- Manipular la base de datos con
dplyroplyr.
- Aesthetics: (aes) para hacer visibles los datos:
x,y: las variables a mapear a las coordenadas \(x,y\).colour: el color de las geometrías de acuerdo a los datos.fill: el color de relleno.group: el grupo al que pertenecerán las geometrías.shape: las formas de los puntos a usar.linetype: el tipo de línea.size: el tamaño de escala.alpha: la transparencia de las geometrías.
- Objetos geométricos: esto determinará el tipo de plot:
geom_point()geom_line()geom_histogram()geom_boxplot()geom_smooth()geom_violin()geom_bar()
- Facetas:
face_wrap()face_grid
- Estadísticos:
- Mostrar medias, summaries.
- Se deben calcular.
- Coordenadas:
coord_cartesiancoord_polarcoord_map
- Themes
- Configuración visual.
- Fuentes, colores, formas.
Vamos a explorar algunas de las geometrías básicas.
x <- c(1,2,3,5,4)
y <- c(2,6,4,3,5)
df <- data.frame(x, y,
label = c("a", "b", "c", "d", "e")
)
p <- ggplot(df, aes(x,y, label = label)) +
labs(x = NULL, y = NULL) +
theme(plot.title = element_text(size = 12))
p + geom_point() +
ggtitle("geom_point()")
Vamos a cargar la base de datos iris y realizar un plot básico con ggplot.
Si observamos, solo hemos creado un plot vacío con los ejes de las variables a mapear. Nos falta indicar el tipo de geometría a usar.
Existen dos formas de poner los elementos aestéticos, una es dentro de la función ggplot y la otra dentro de las funciones geom_function().
¿Cuál es la diferencia? Si solo contamos con una capa como en estos dos casos, no vamos a notar una diferencia. Como mencionamos antes, ggplot trabaja con capas y las características de los aes dependen de en que capa se coloquen.
Por ejemplo, con los temas o colores:
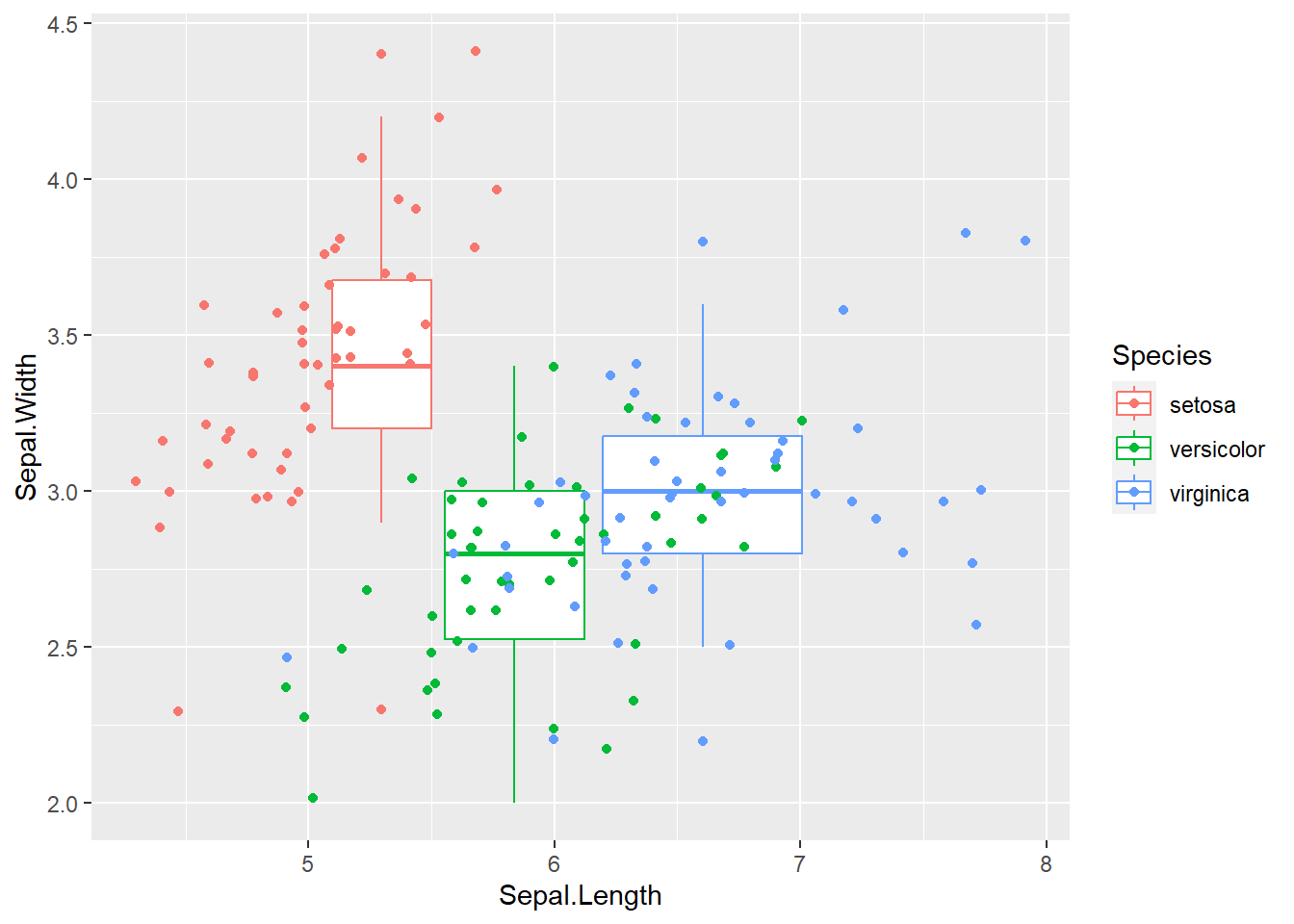
ggplot(iris) +
aes(x=Sepal.Length,y=Sepal.Width, group = Species) +
geom_boxplot() +
geom_jitter(aes(colour = Species))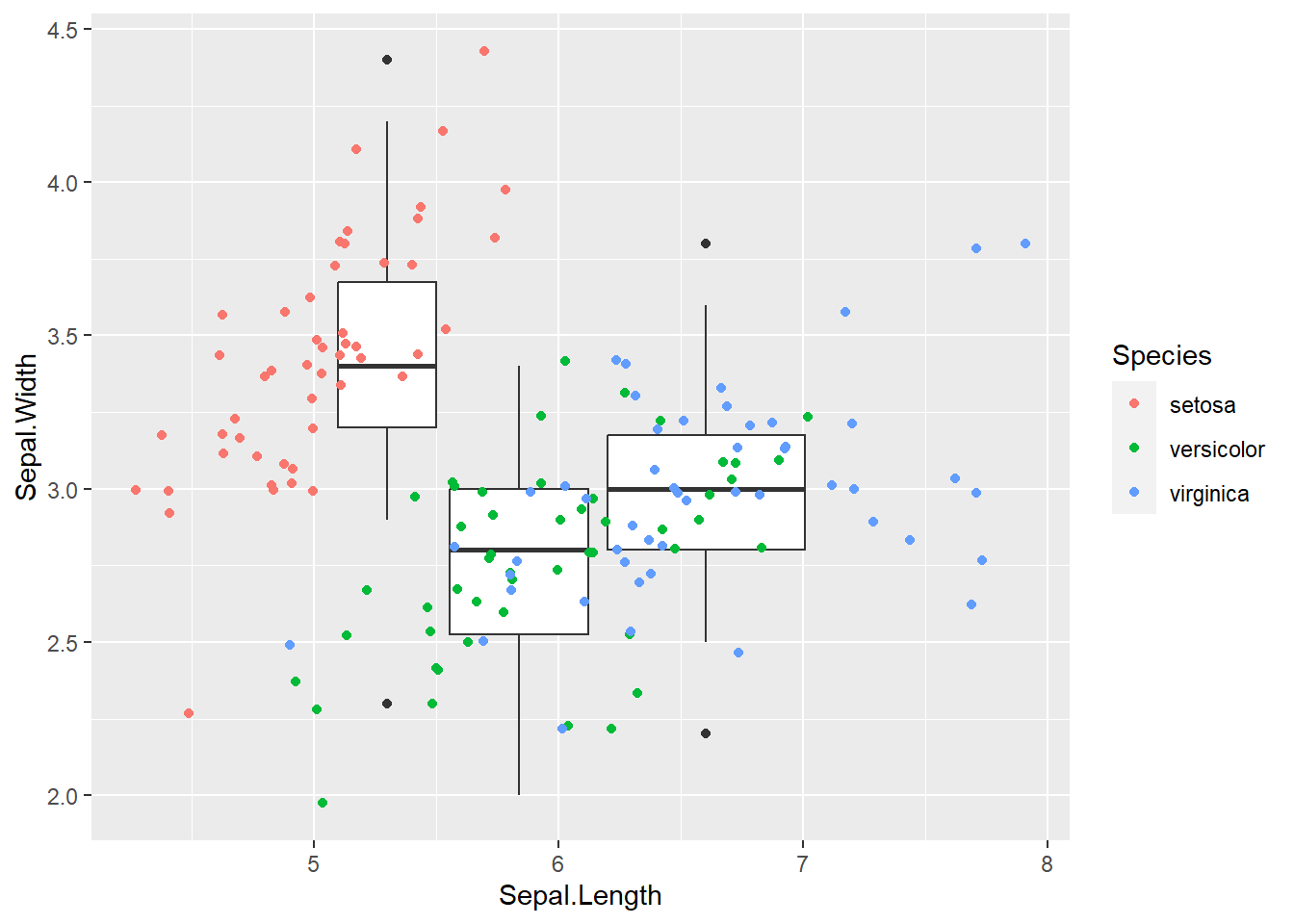
Con los plots en ggplot debemos tener cuidado en como usar las variables continuas y discretas para los aes
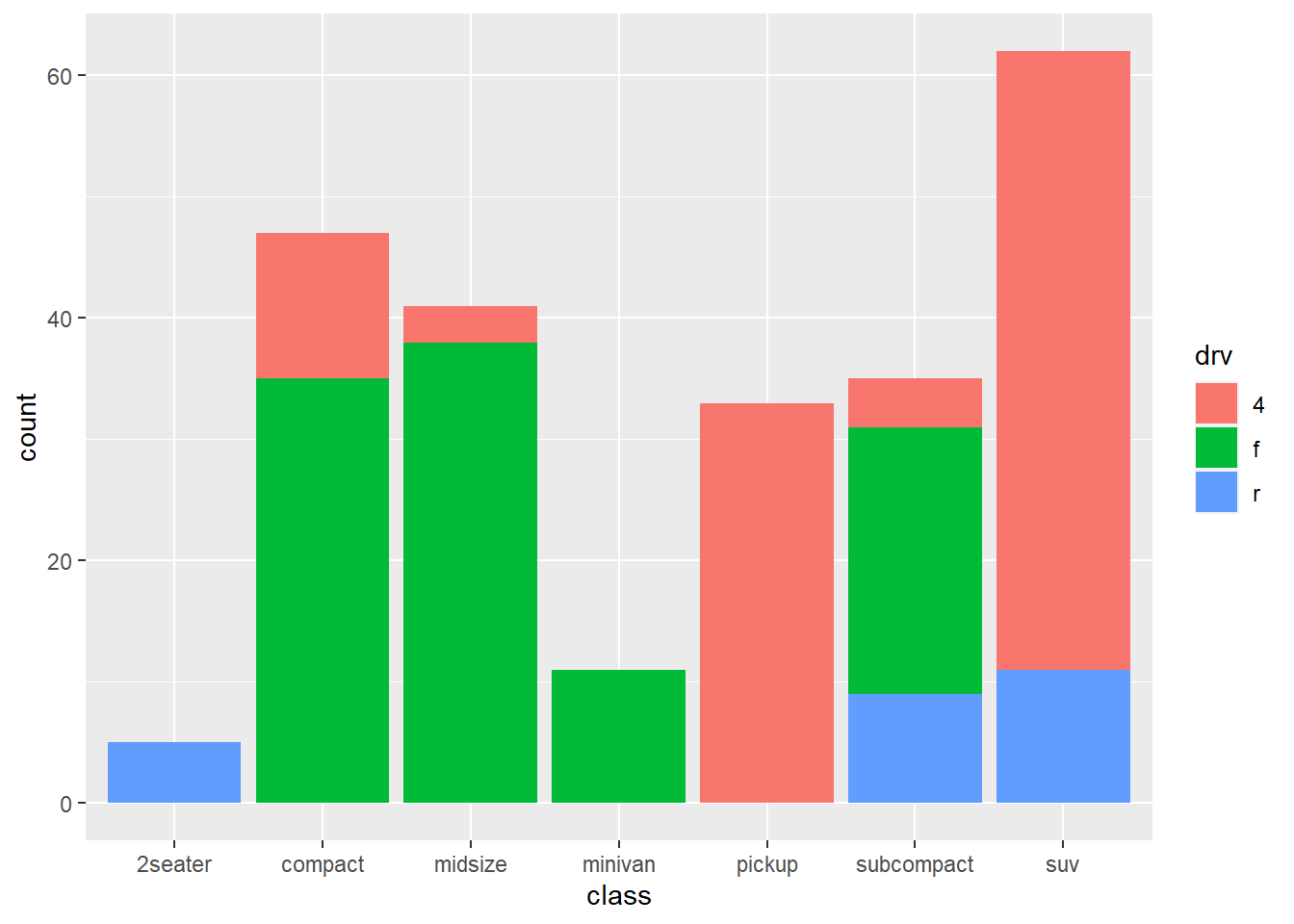
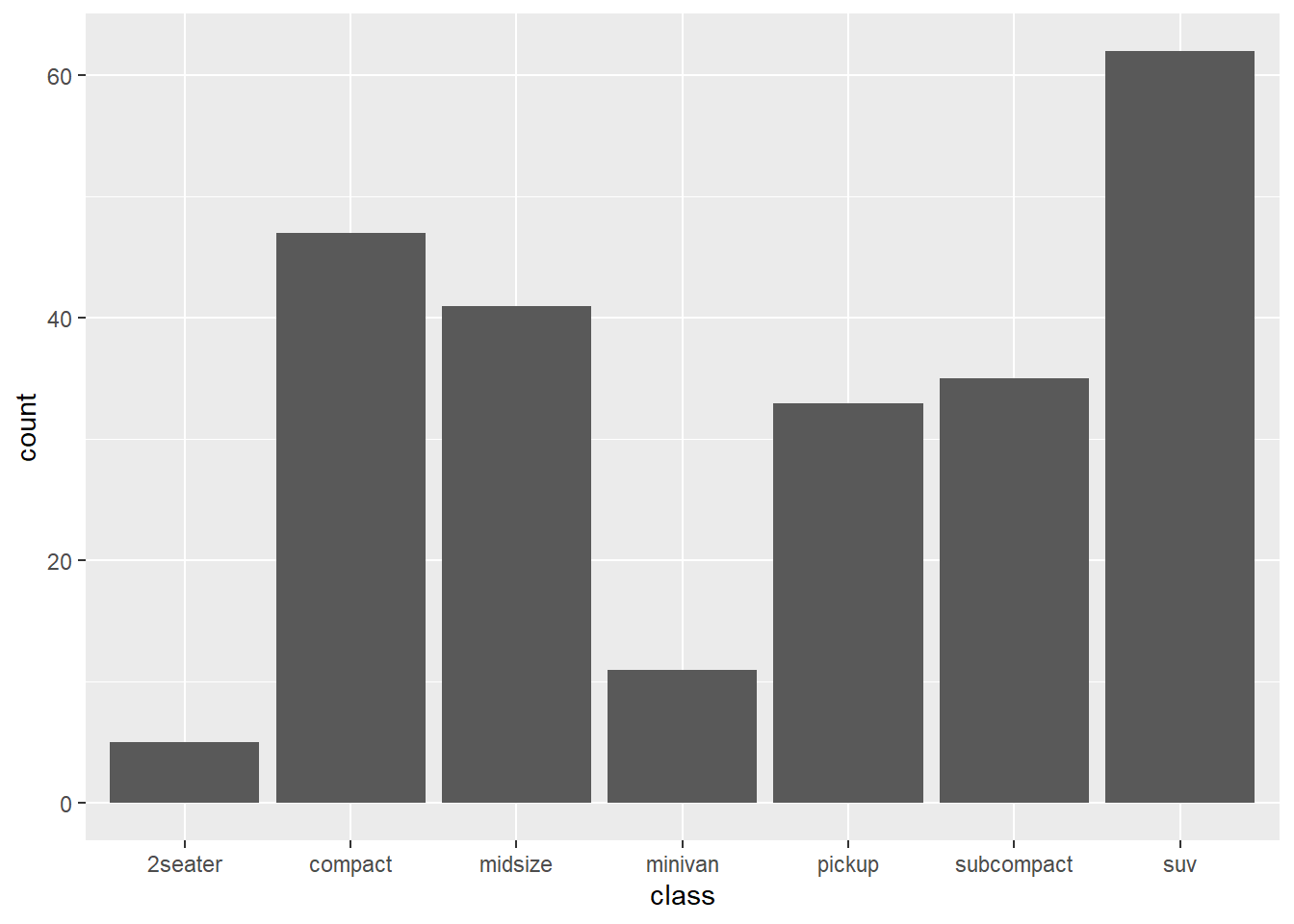
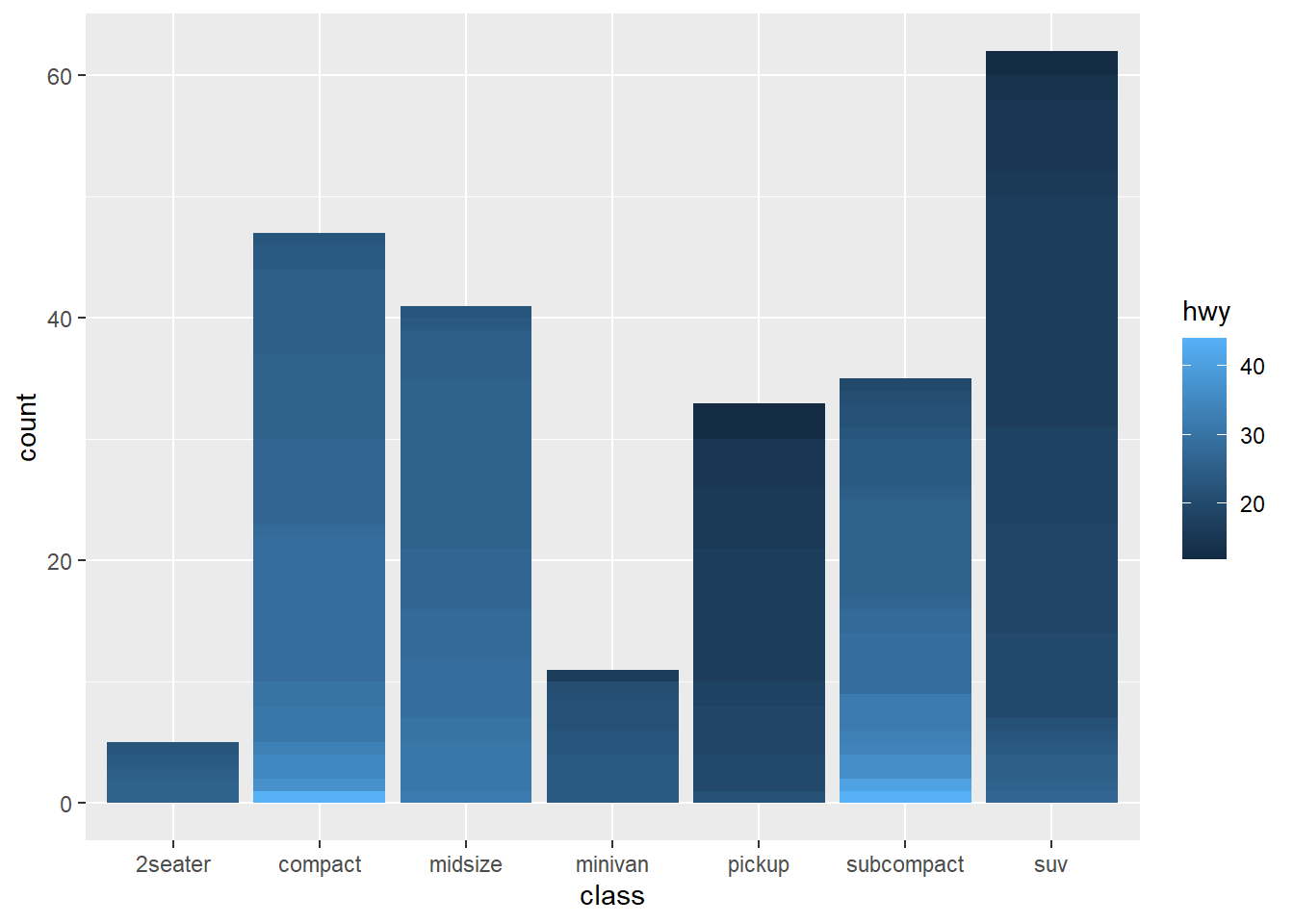
En estos gráficos también podemos agrefar ciertos resumenes estadísticos dependiendo del tipo de variable que tengamos, continuas o discretas.
y <- c(18, 11, 16)
df <- data.frame(x = 1:3, y = y, se = c(1.2, 0.5, 1.0))
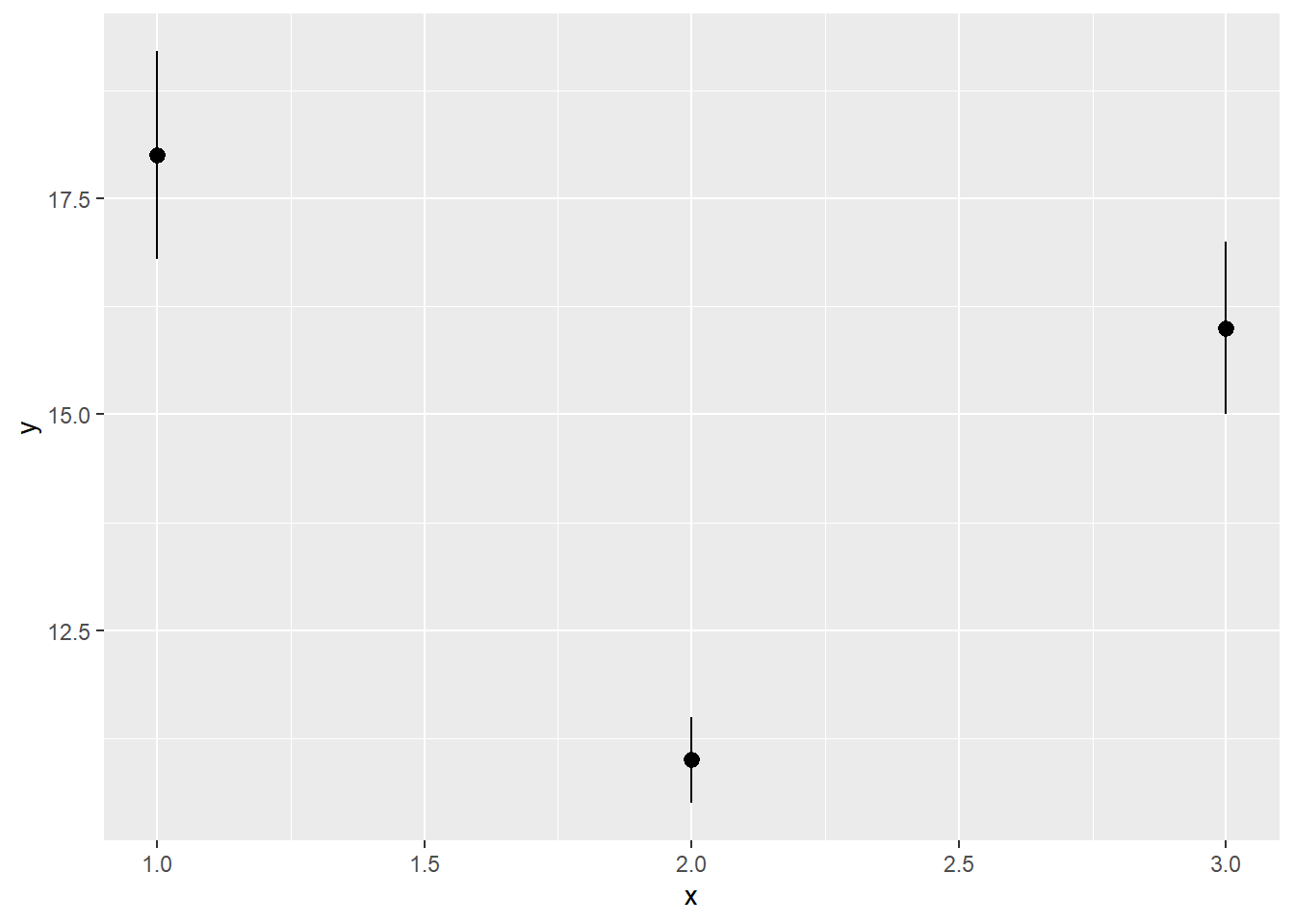
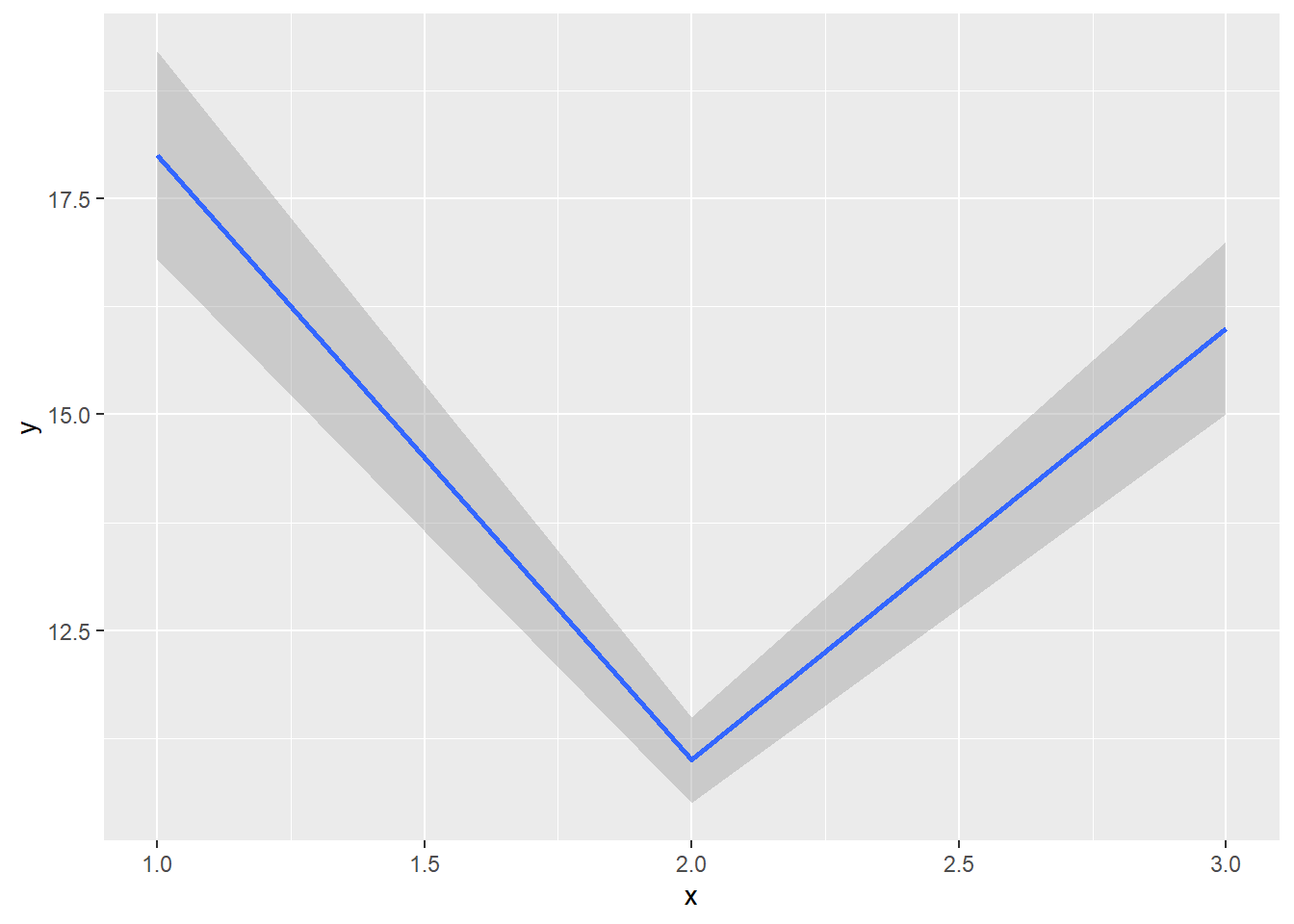
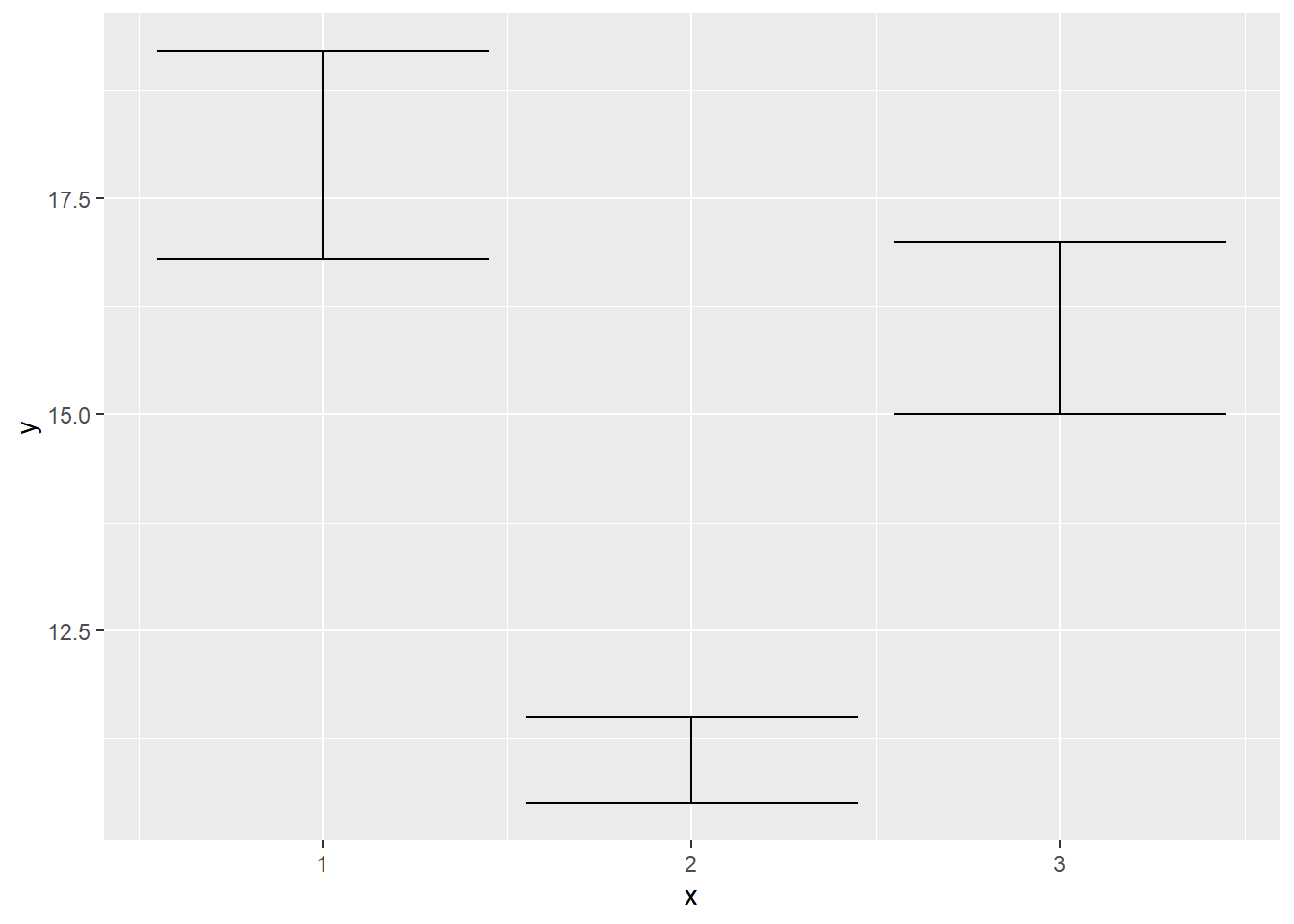
base <- ggplot(df, aes(x, y, ymin = y - se, ymax = y + se))
base + geom_crossbar()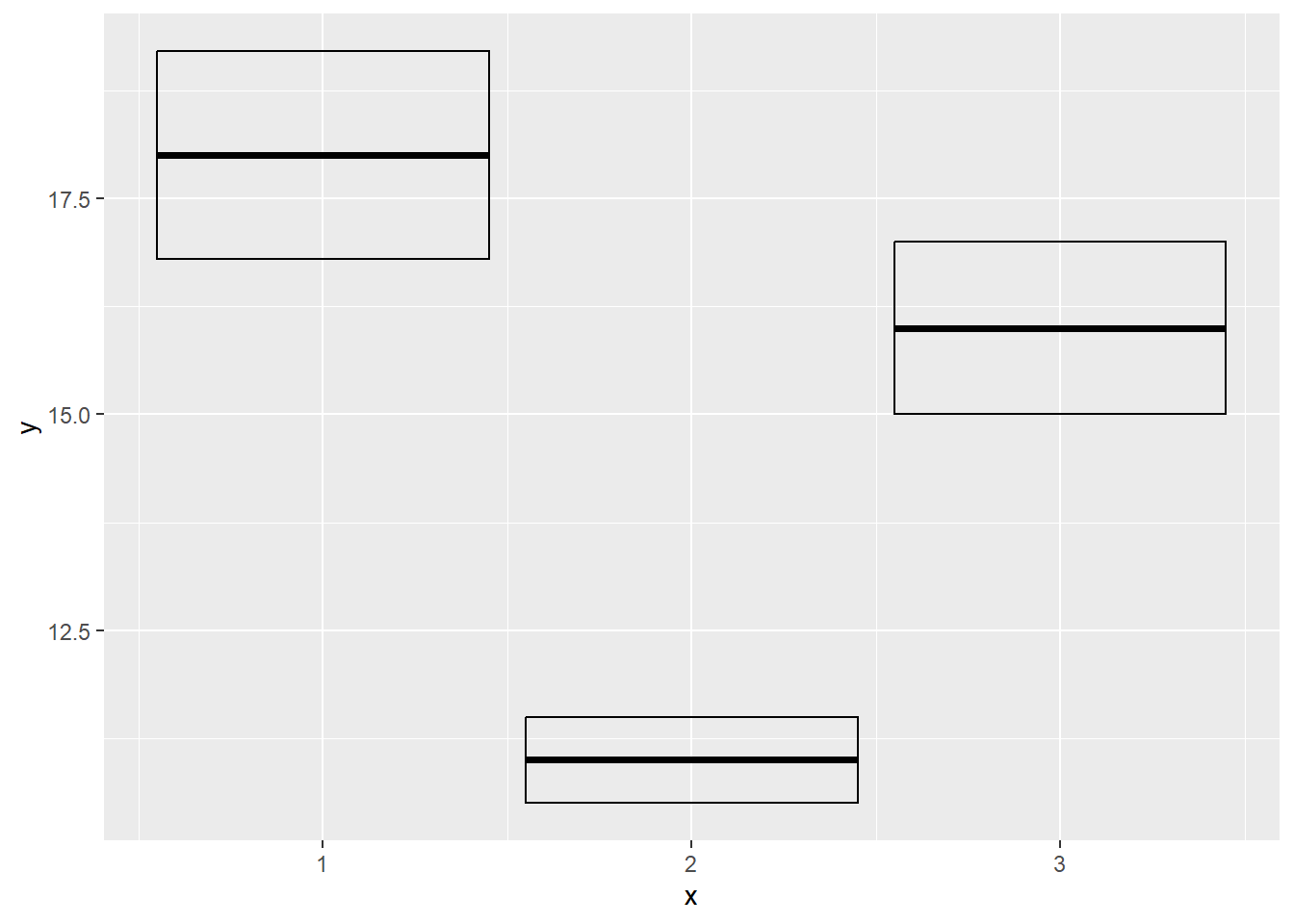
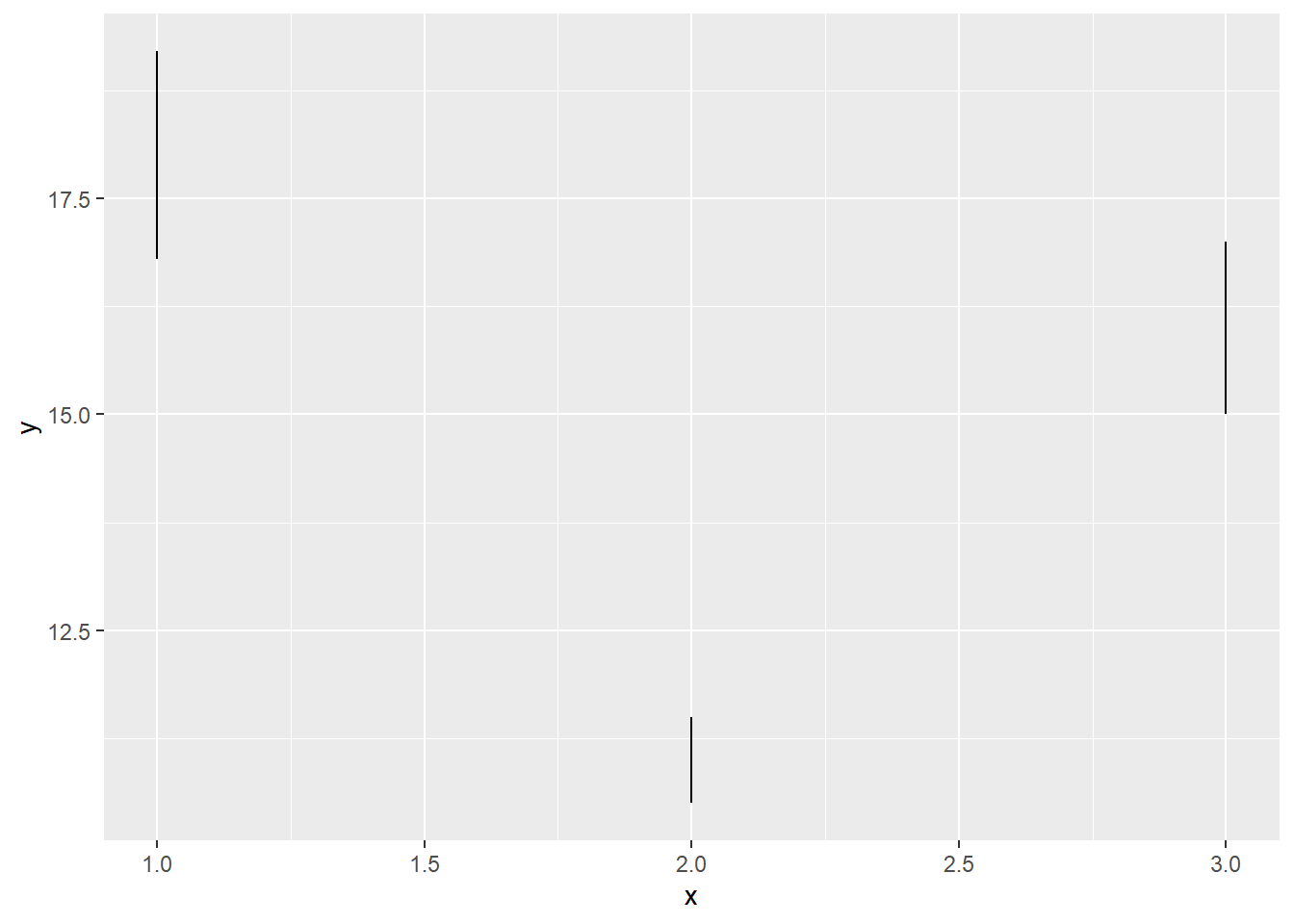
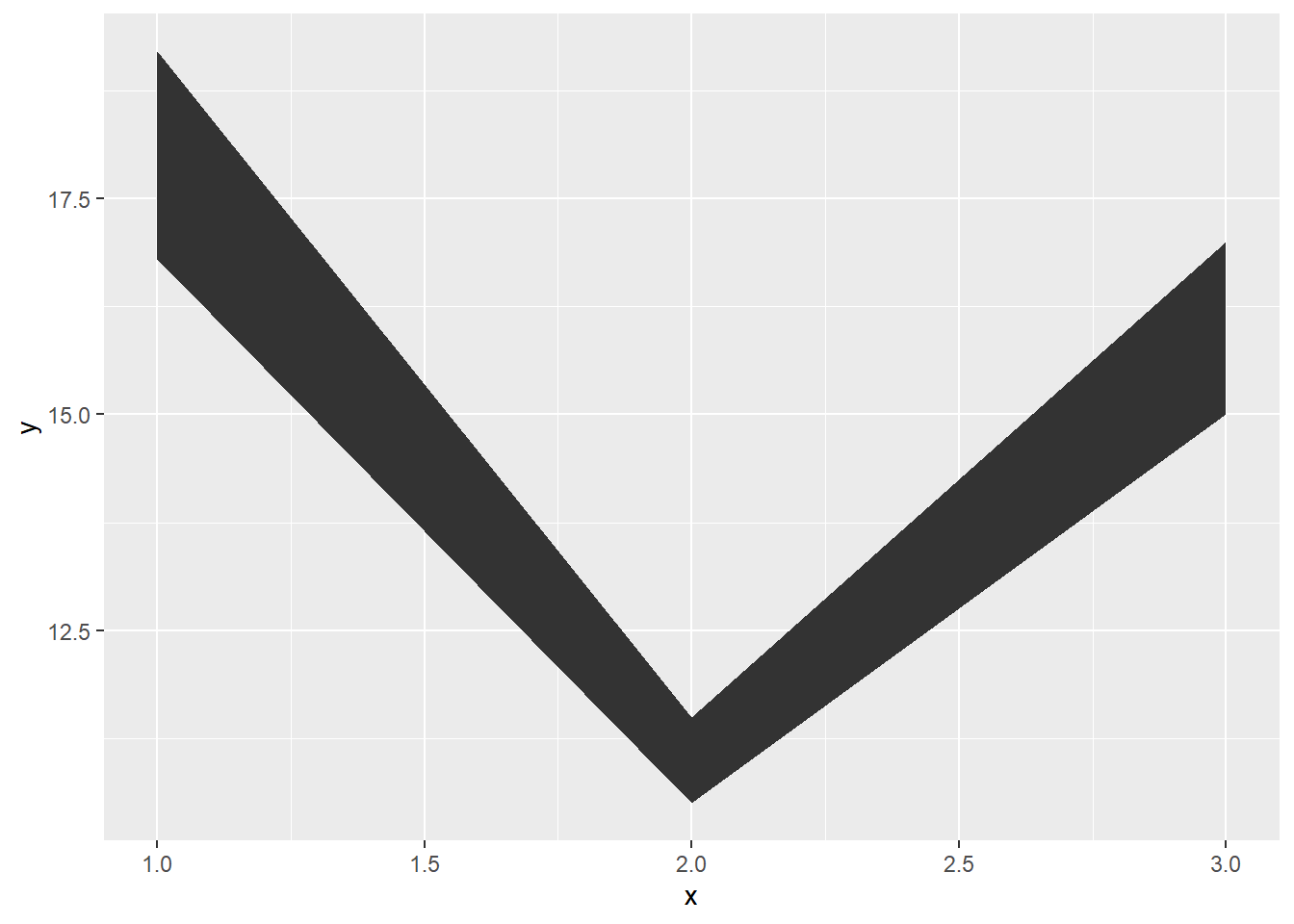
En ocasiones tenemos datos con peso, es decir tenemos múltiples observaciones por línea y nos interesa en este caso darle un peso a dichas variables. Vamos a cargar la base de datos midwest.
data("midwest")
# Pobalción sin peso
ggplot(midwest, aes(percwhite, percbelowpoverty)) +
geom_point()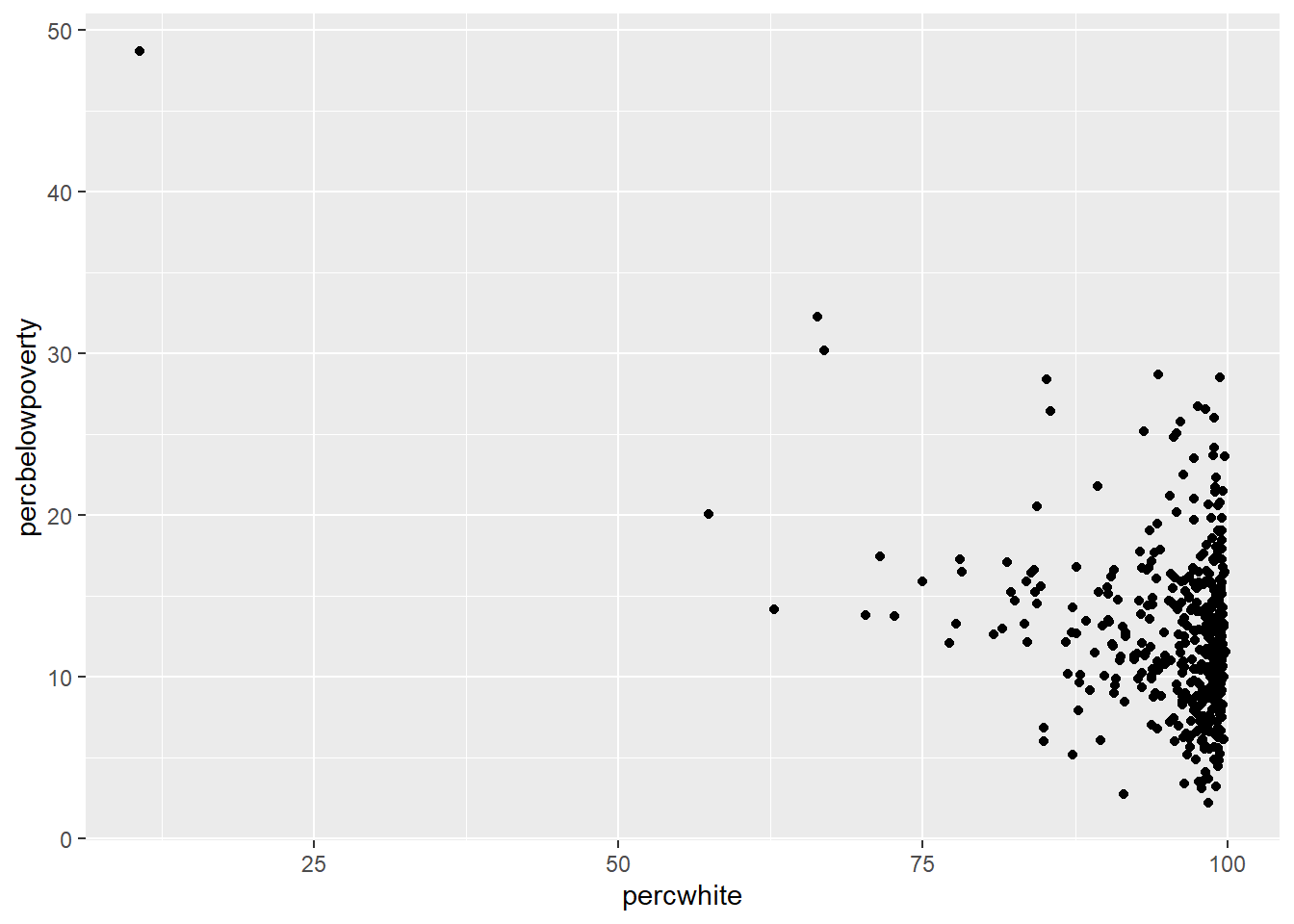
# Población con peso
ggplot(midwest, aes(percwhite, percbelowpoverty)) +
geom_point(aes(size = poptotal / 1e6)) +
scale_size_area("Population\n(millions)", breaks = c(0.5, 1, 2, 4))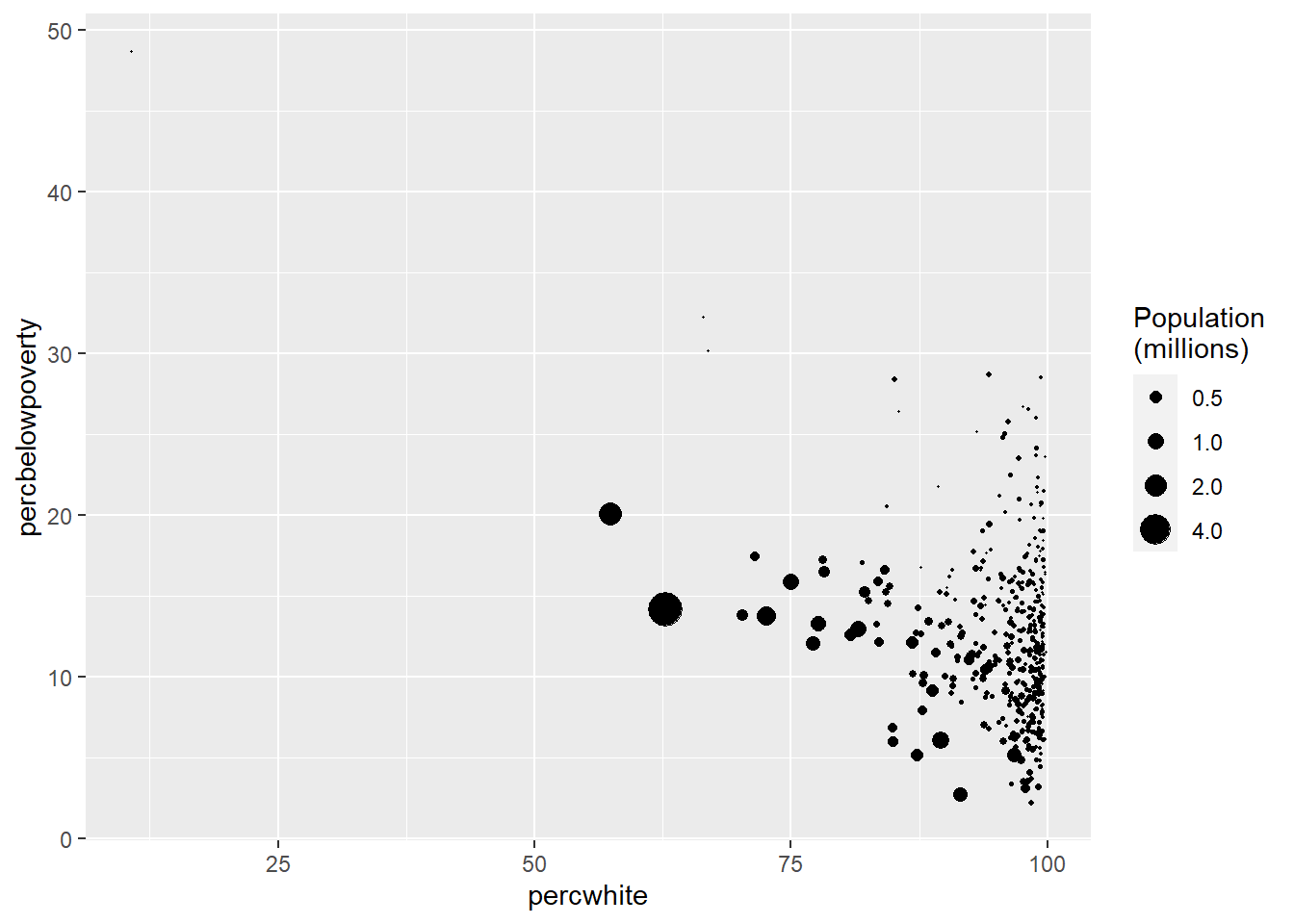
Un ejemplo de como podemos usar los resúmenes estadísticos es el siguiente. En este caso estamos usando un weight en los aestéticos de diferente forma.
# Sin peso
ggplot(midwest, aes(percwhite, percbelowpoverty)) +
geom_point() +
geom_smooth(method = lm, linewidth = 1)## `geom_smooth()` using formula = 'y ~ x'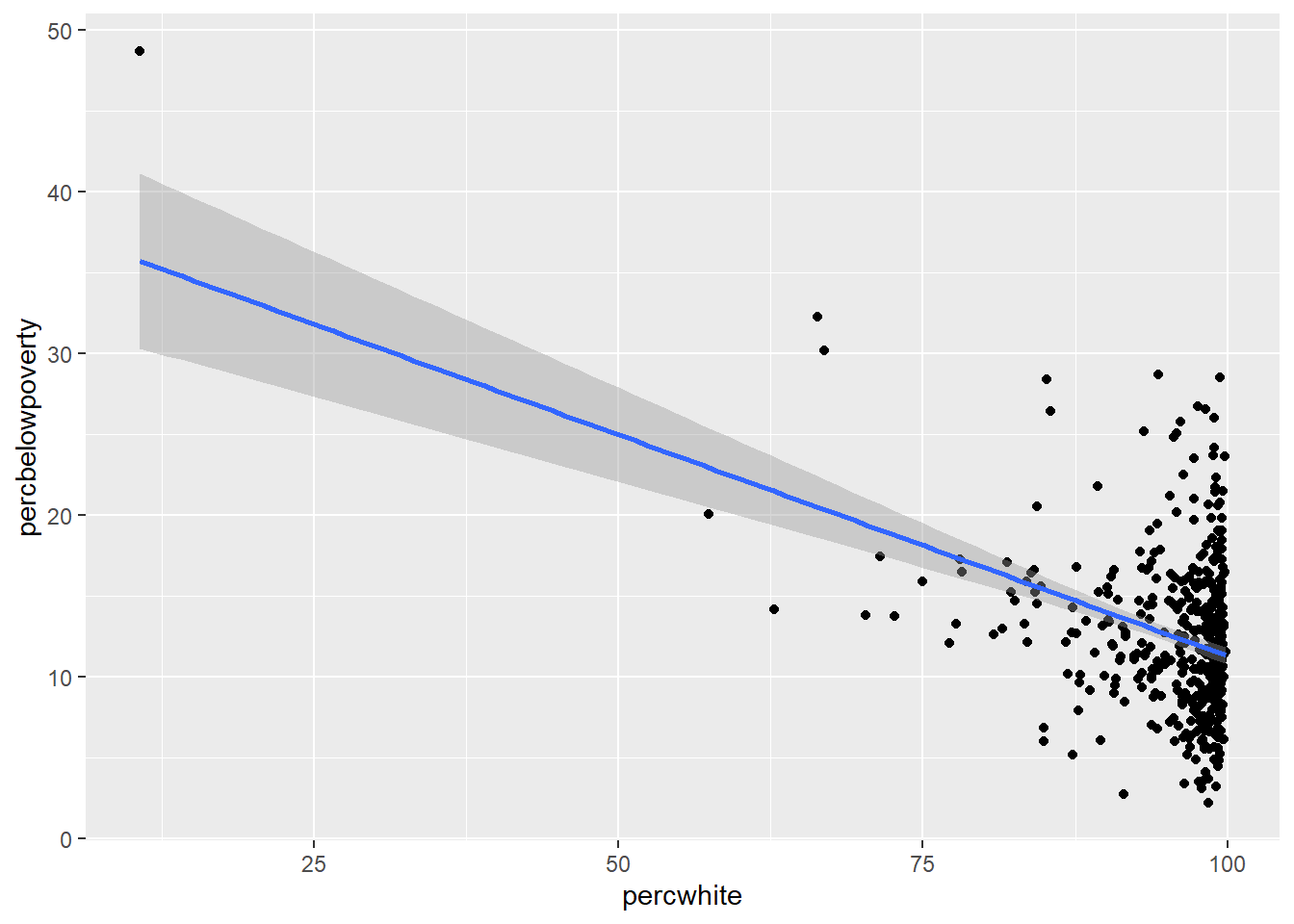
# Con peso
ggplot(midwest, aes(percwhite, percbelowpoverty)) +
geom_point(aes(size = poptotal / 1e6)) +
geom_smooth(aes(weight = poptotal), method = lm, linewidth = 1) +
scale_size_area(guide = "none")## `geom_smooth()` using formula = 'y ~ x'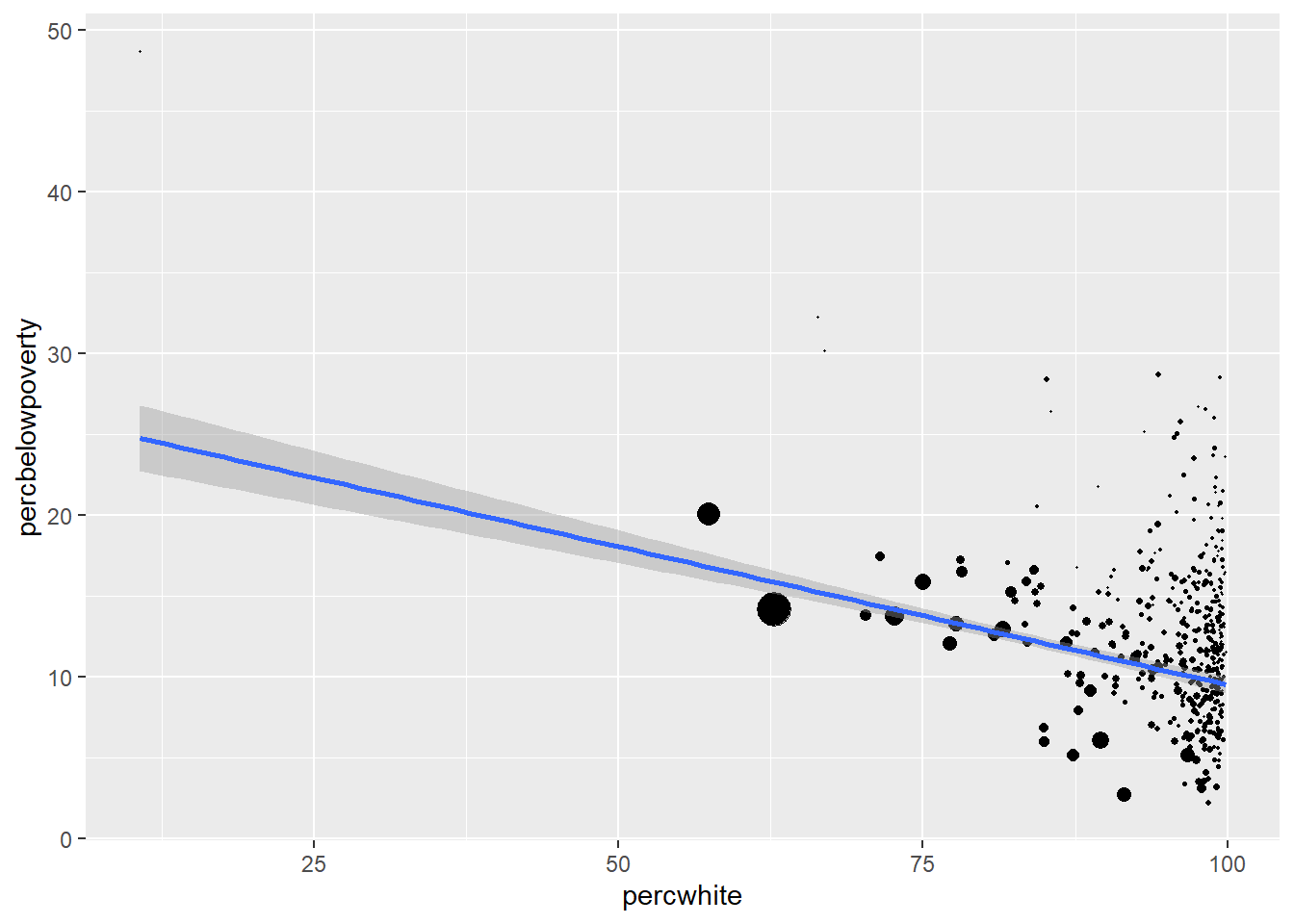
En el caso de los histogramas, cuando agregamos las opciones de peso pasaríamos de por ejemplo un histograma que represente la distribución de la frecuencia por el del número de población.
ggplot(midwest, aes(percbelowpoverty)) +
geom_histogram(aes(weight = poptotal), binwidth = 1) +
ylab("Población (1000)")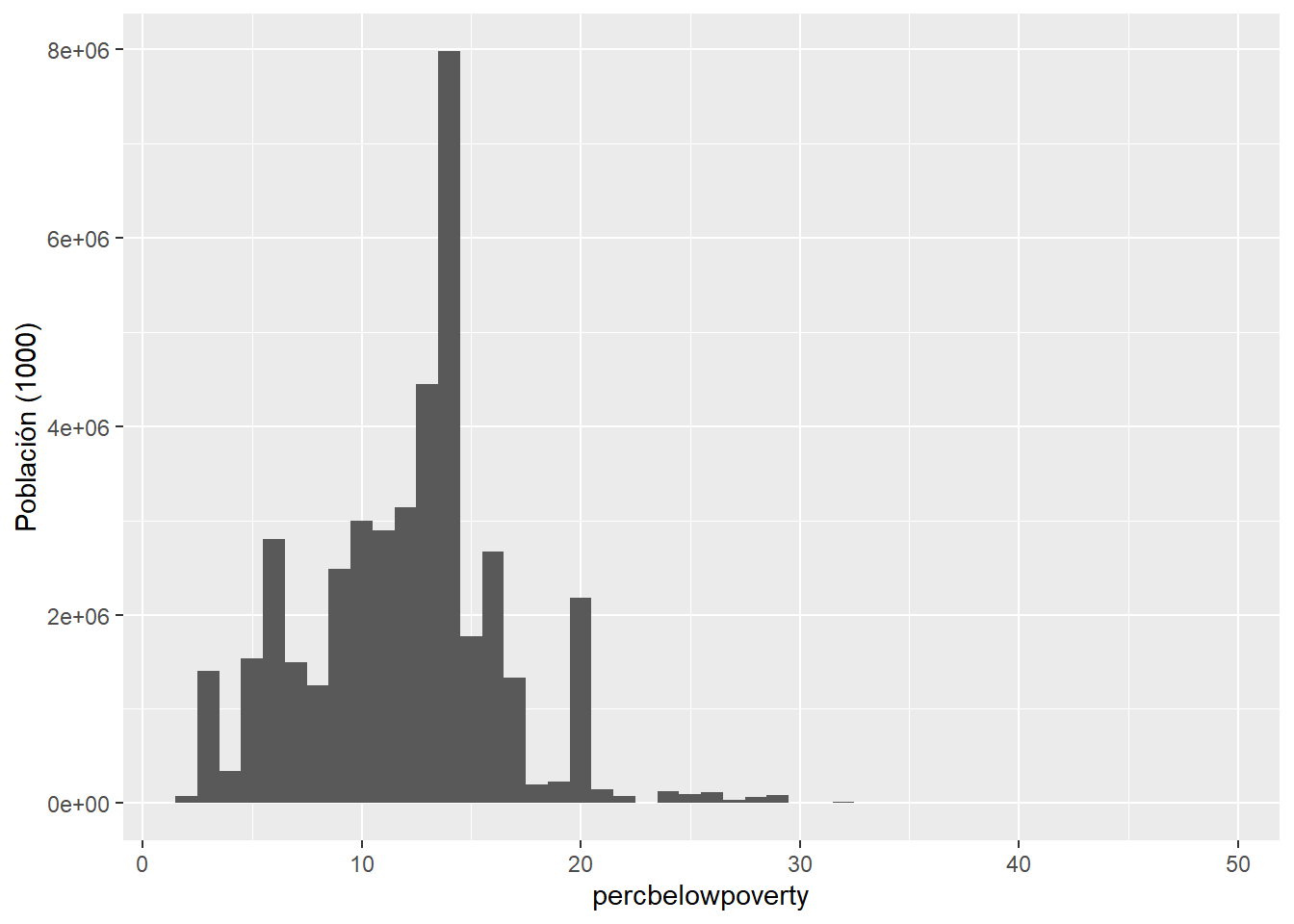 Vamos a empezar a manipular varias capas. Podemos modificar colores y formas al especificarlo en los aestéticos de la función de geometría a usar.
Vamos a empezar a manipular varias capas. Podemos modificar colores y formas al especificarlo en los aestéticos de la función de geometría a usar.
ggplot(iris, aes(x=Sepal.Length, y=Sepal.Width)) +
geom_jitter(aes(colour = Species, shape = Species), size = 2.0, alpha = 0.8)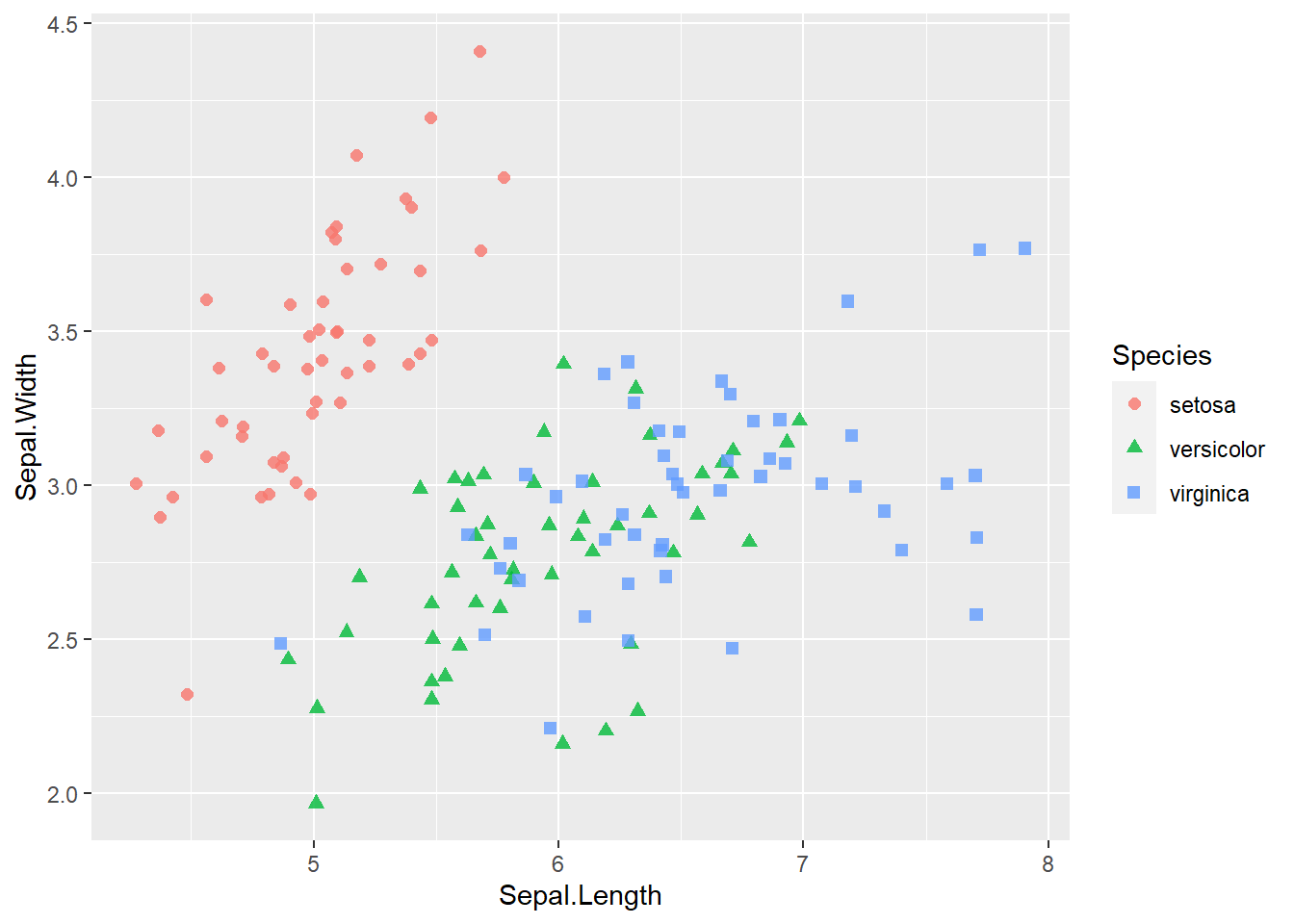
Podemos cambiar la paleta de colores y formas:
ggplot(iris, aes(x=Sepal.Length, y=Sepal.Width)) +
geom_jitter(aes(colour = Species, shape = Species), size = 2.0, alpha = 0.8) +
scale_shape_manual(values = c(17,18,19)) +
scale_color_manual(values = c("#FF33CC", "#CC66FF", "#33C9CC"))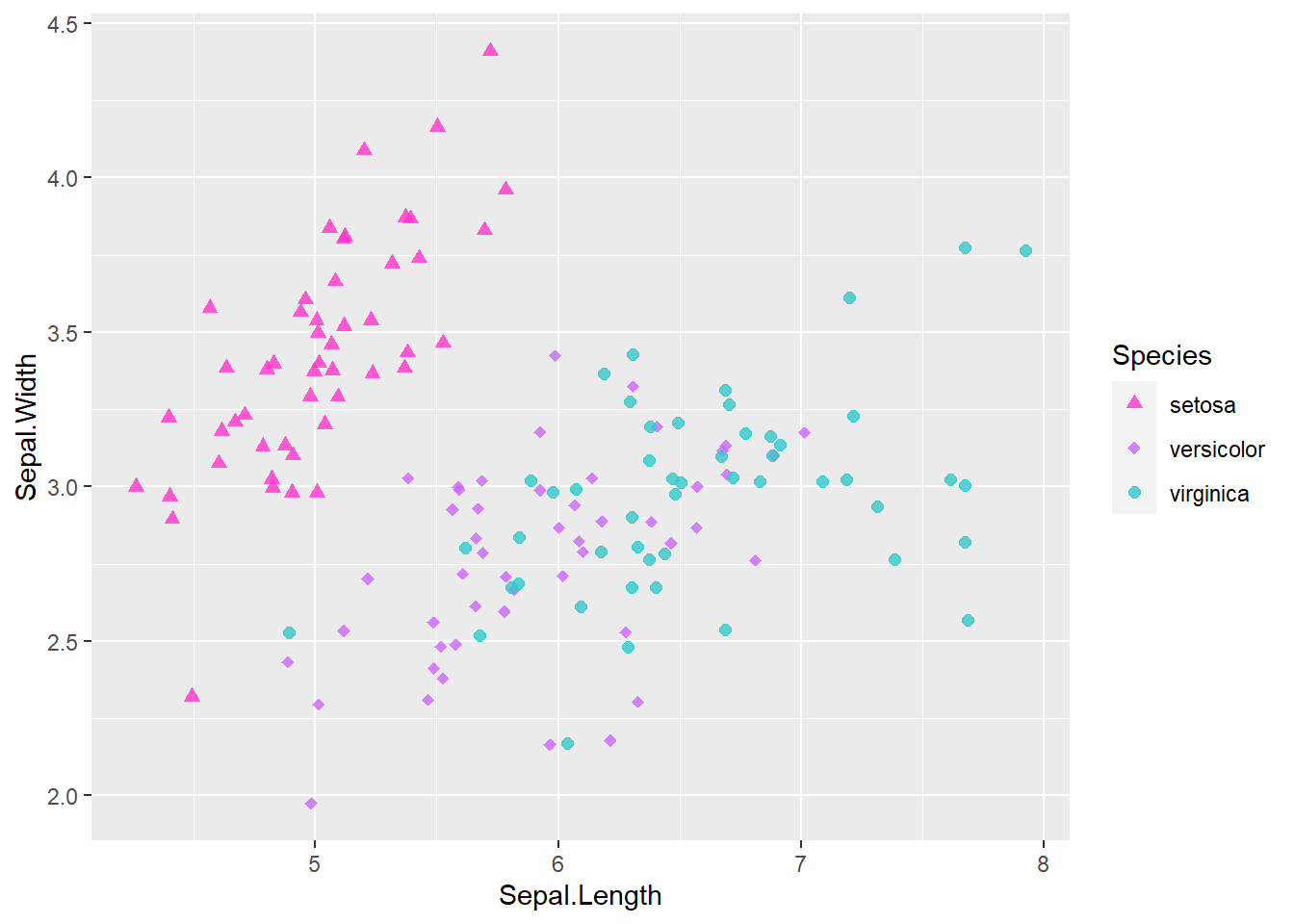
Podemos cambiar títulos y ejes, agregar captions, legendas.
p <- ggplot(iris, aes(x=Species, y=Sepal.Width, fill = Species)) +
geom_boxplot() +
geom_jitter(colour = "gray") +
labs(title = "Rango del Ancho Sepal",
subtitle = "Iris data set",
x = "Especies",
y = "Ancho Sepal",
caption = "Datos: Iris data set from `data(iris)`",
fill = "Especies")
p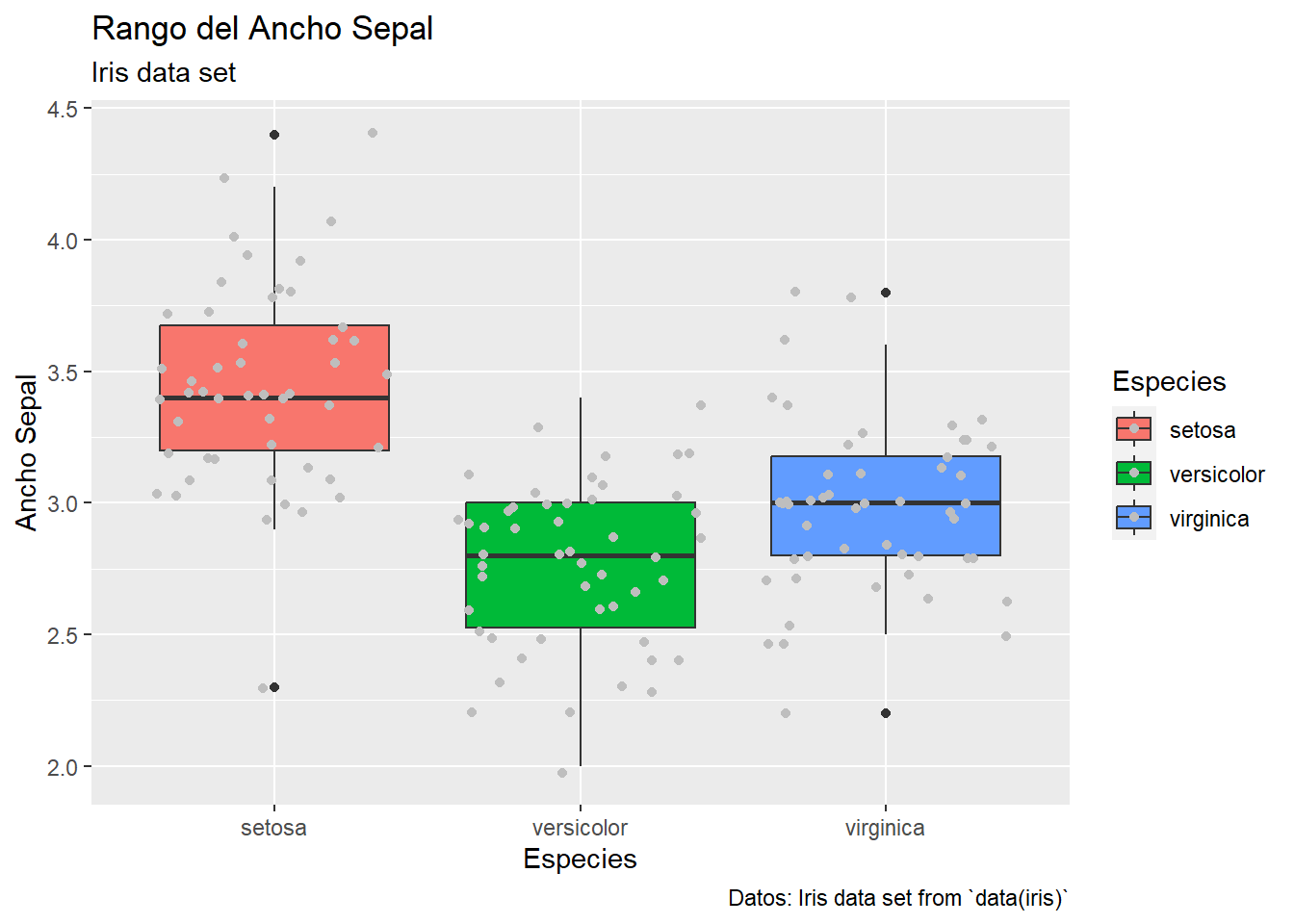
Existen varios temas que se pueden usar, algunos los pueden encontrar en esta liga.
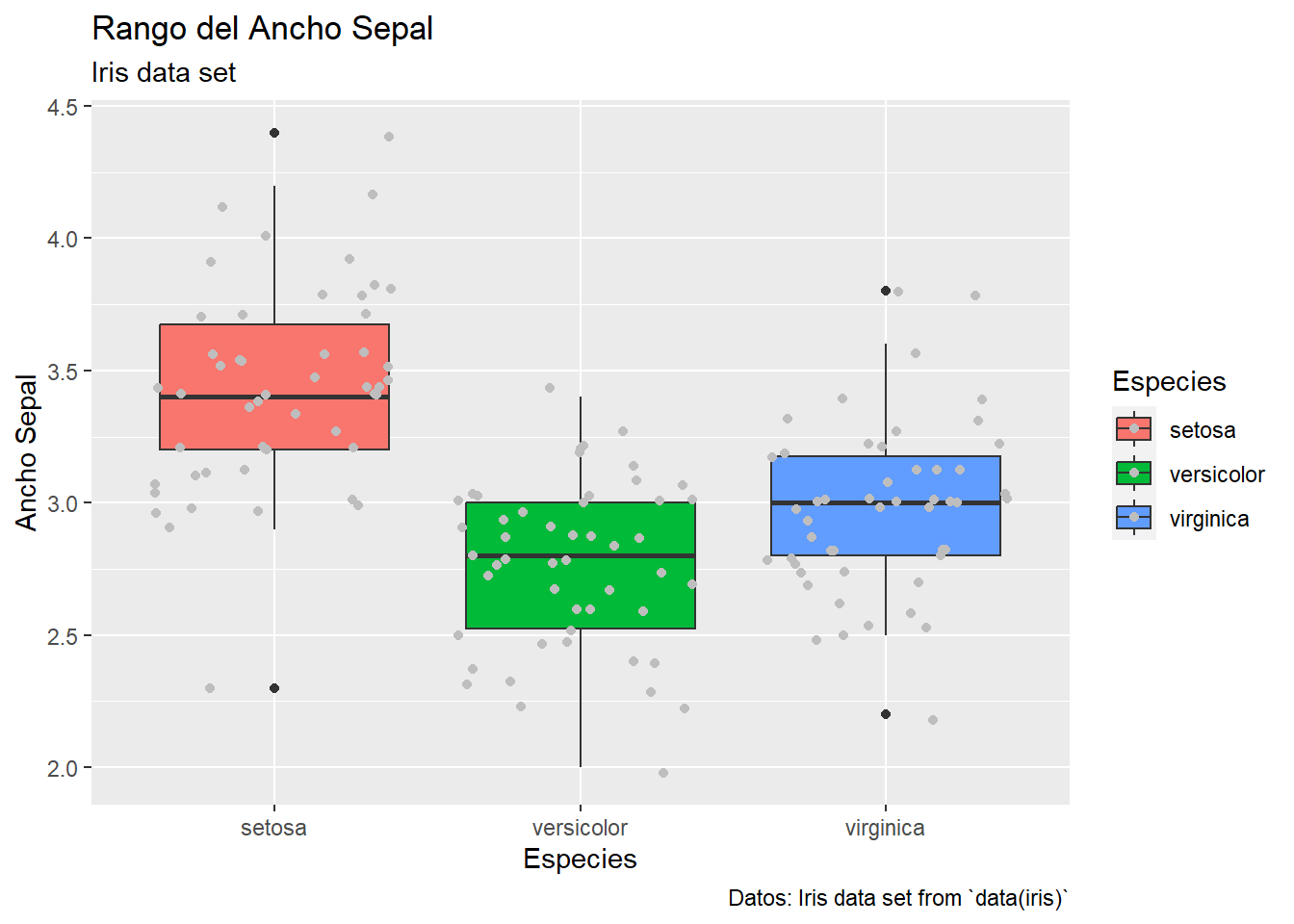
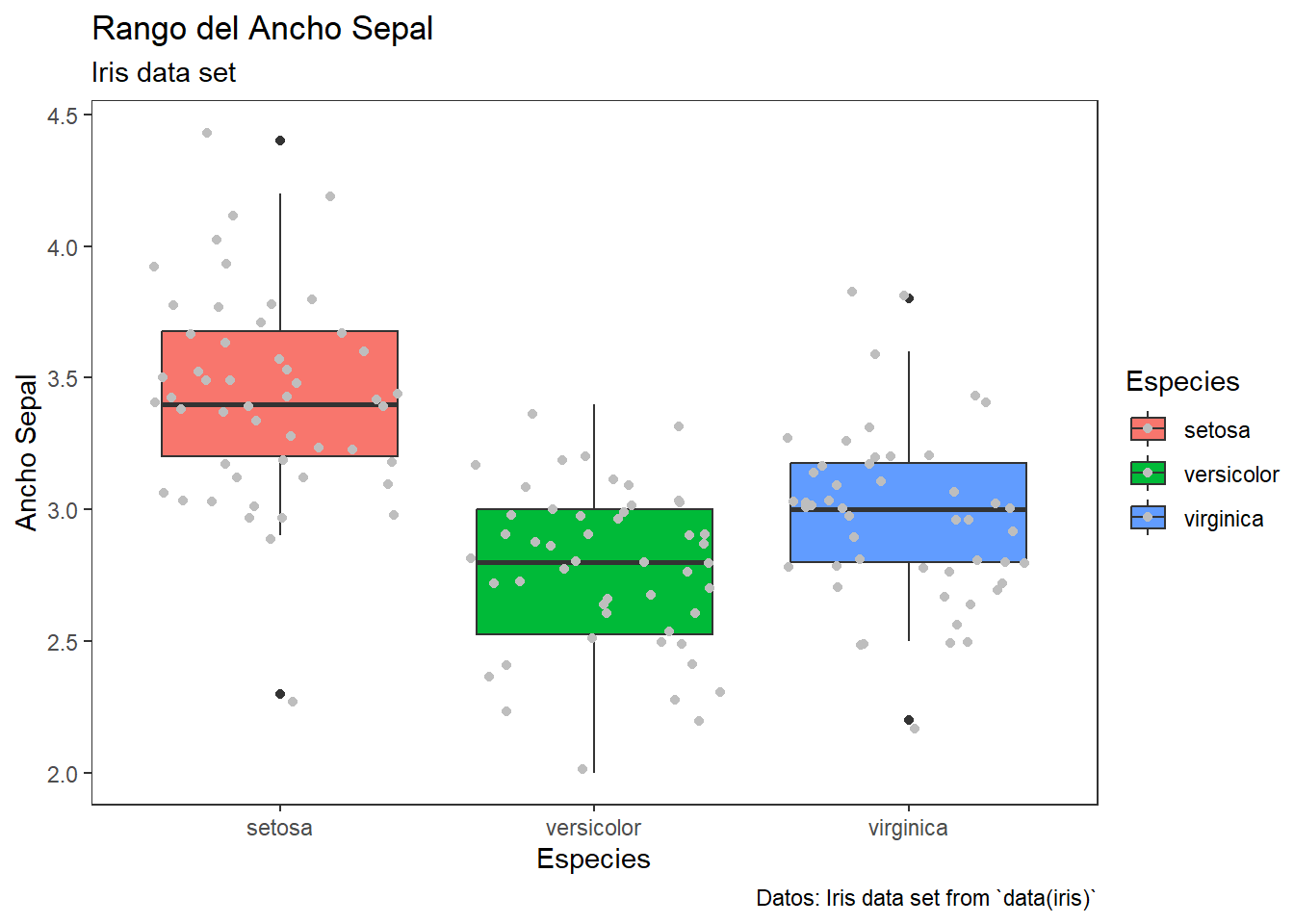
También podemos modifiar el estilo de letras y ejes.
p + theme(plot.title = element_text(color = "red", size = 12, face = "bold.italic"),
axis.title.x = element_text(color = "blue", size = 10, face = "bold"),
axis.title.y = element_text(color = "#993333", size = 10, face = "italic"),
axis.text = element_text(color = "slateblue2", size = 10))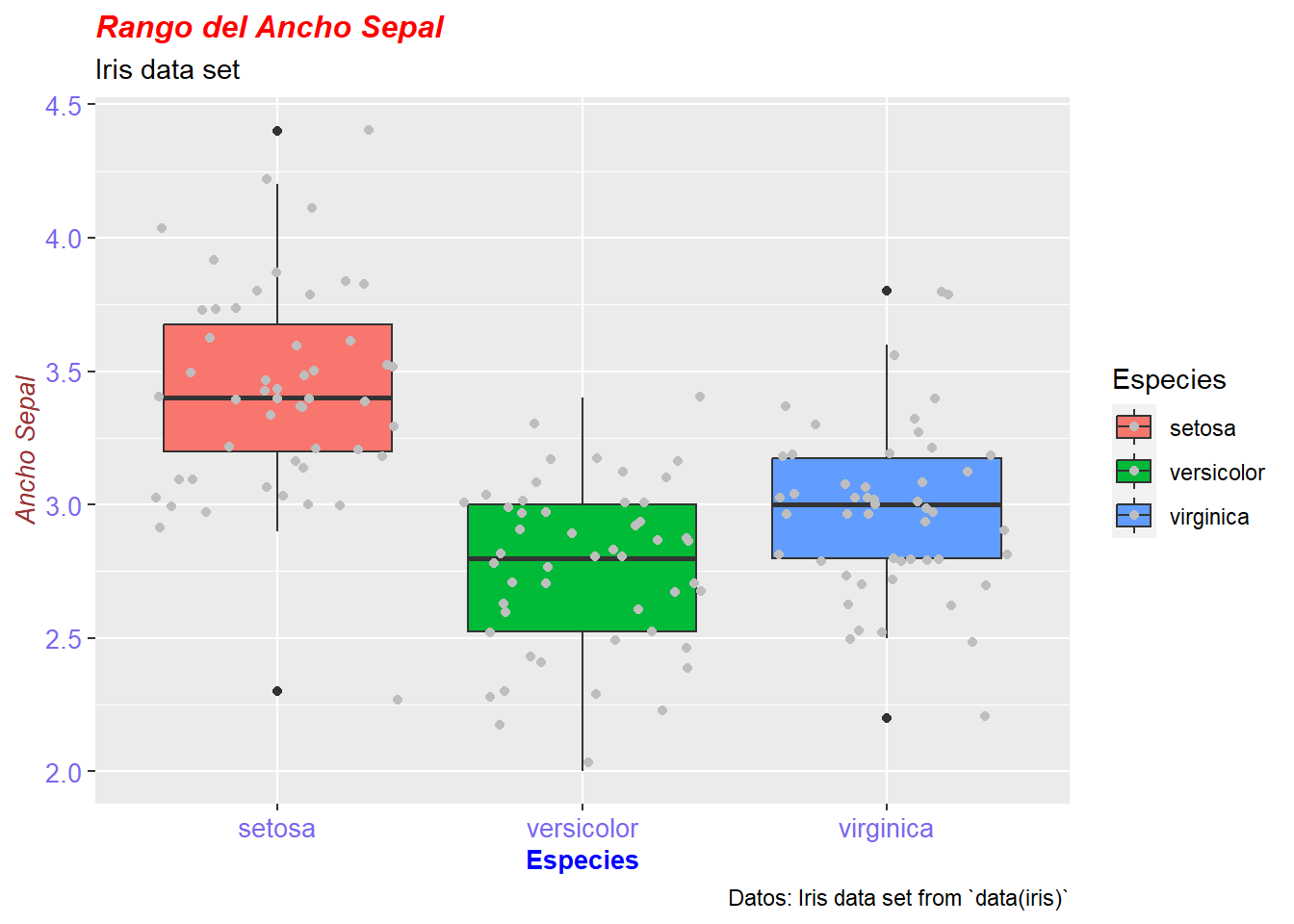
p + theme_dark() +
theme(plot.title = element_text(color = "red", size = 12, face = "bold.italic"),
axis.title.x = element_text(color = "blue", size = 10, face = "bold"),
axis.title.y = element_text(color = "#993333", size = 10, face = "italic"),
axis.text = element_text(color = "slateblue2", size = 10))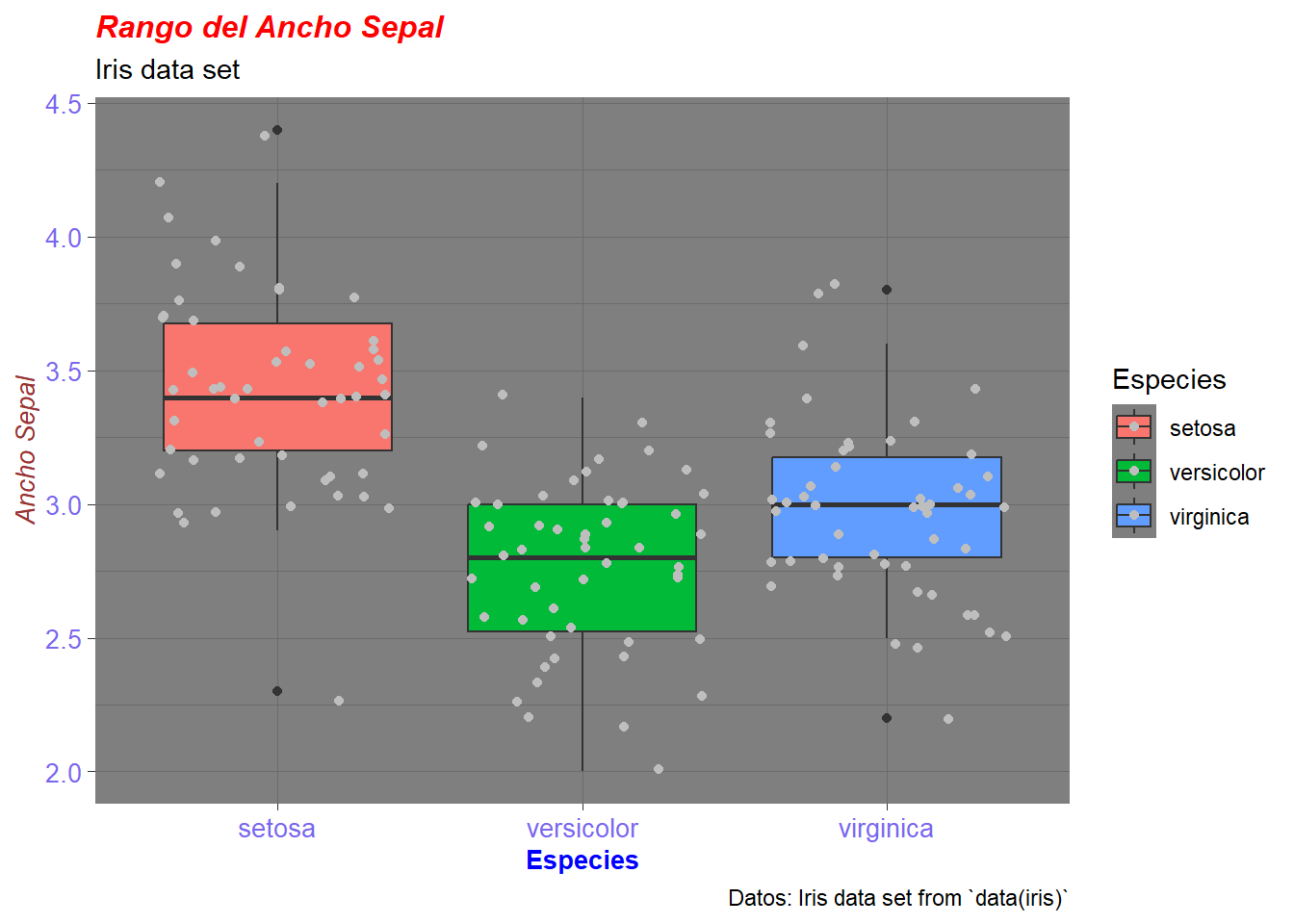
Podemos crear paletas manuales o usar algunas paletas ya predeterminadas.
cl <- c("maroon3", "sienna2", "skyblue1")
p <- ggplot(iris, aes(x=Sepal.Length, y=Sepal.Width))+
geom_point(aes(colour = Species)) +
scale_colour_manual(values=cl)+
labs(title = "Longitud y Ancho Sepal",
subtitle = "Iris data set",
x = "Longitud Sepal",
y = "Ancho Sepal",
caption = "Datos: Iris data set from `data(iris)`",
fill = "Especies")
p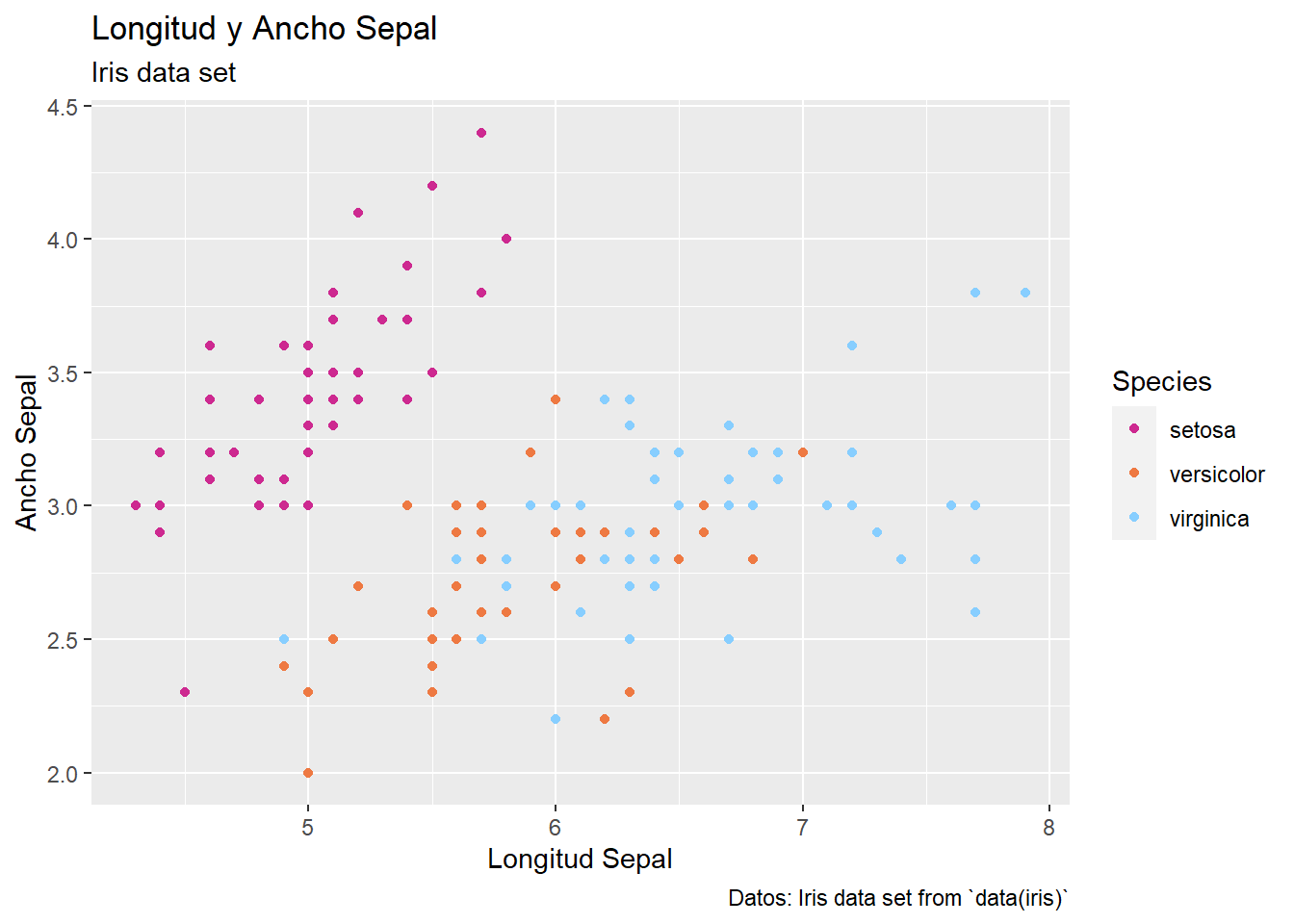
cl2 <- c(colors()[105], colors()[20] , colors()[500])
p <- ggplot(iris, aes(x=Species, y=Sepal.Width, fill = Species))+
geom_bar(stat = "identity") +
scale_fill_manual(values=cl2, labels = c("setosa", "versicolor", "virginica"))+
labs(title = "Rango del Ancho Sepal",
subtitle = "Iris data set",
x = "Especies",
y = "Ancho Sepal",
caption = "Datos: Iris data set from `data(iris)`",
fill = "Especies")
p
library(RColorBrewer)
p <- ggplot(iris, aes(x=Sepal.Length, y=Sepal.Width))+
geom_point(aes(colour = Species)) +
scale_colour_brewer(palette = 'Set1')+
labs(title = "Longitud y Ancho Sepal",
subtitle = "Iris data set",
x = "Longitud Sepal",
y = "Ancho Sepal",
caption = "Datos: Iris data set from `data(iris)`",
fill = "Especies")
p
Existe otra paleta de colores que se llama paletteer y tiene varias escalas continuas de colores.
#install.packages("ggthemes")
#install.packages("paletteer")
library(paletteer)
data(airquality)
ggplot(airquality, aes(Temp, Wind, colour = Wind)) +
geom_point() +
scale_colour_gradientn(colours=rainbow(30))
p1<-paletteer_c("ggthemes::Blue-Green Sequential", 30)
ggplot(airquality, aes(Temp, Wind, colour = Wind)) +
geom_point() +
scale_colour_gradientn(colours=p1)
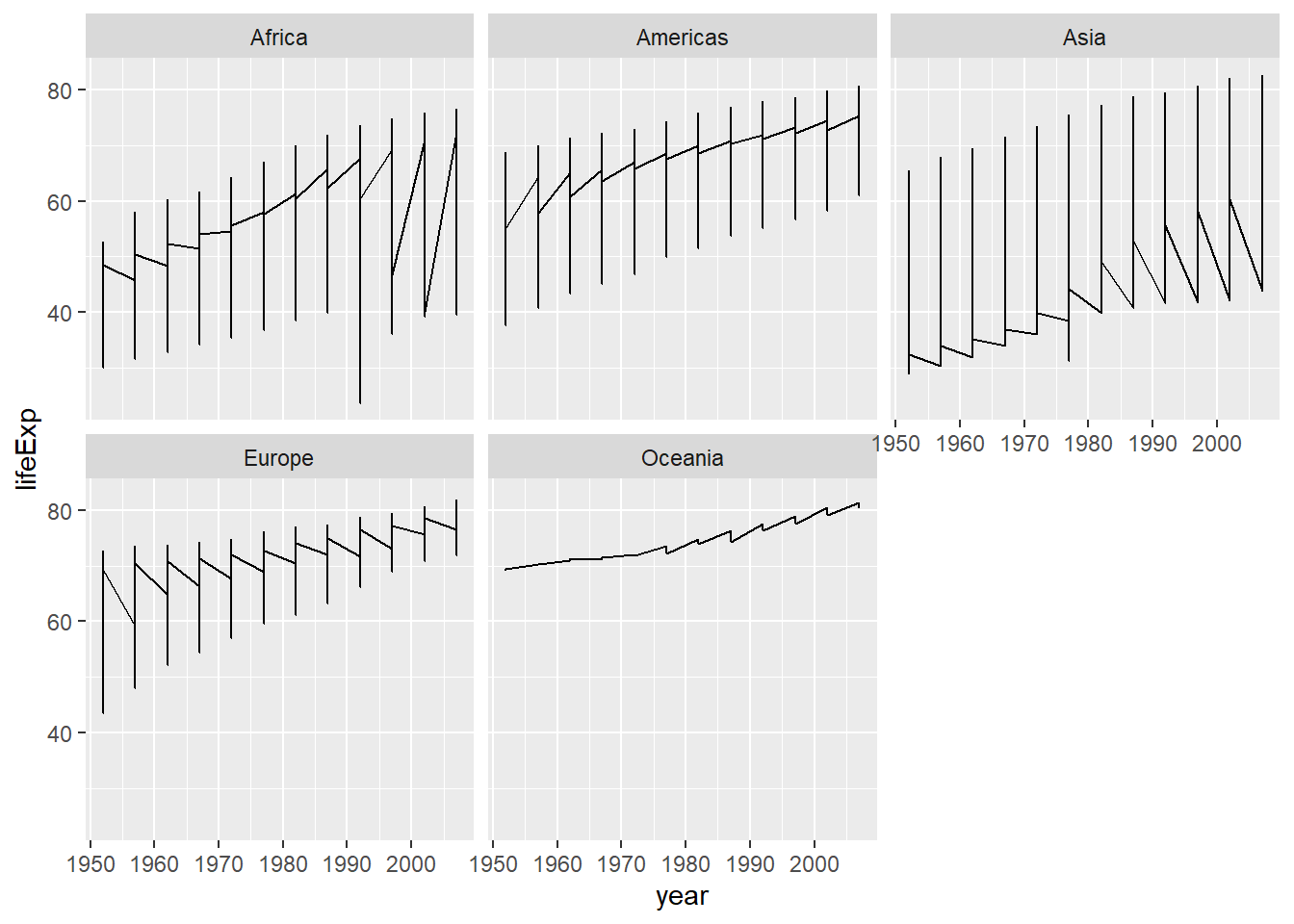
En ocasiones queremos tener como un grid de varios plots, para eso ocupamos facet_grid o facet_wrap.
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.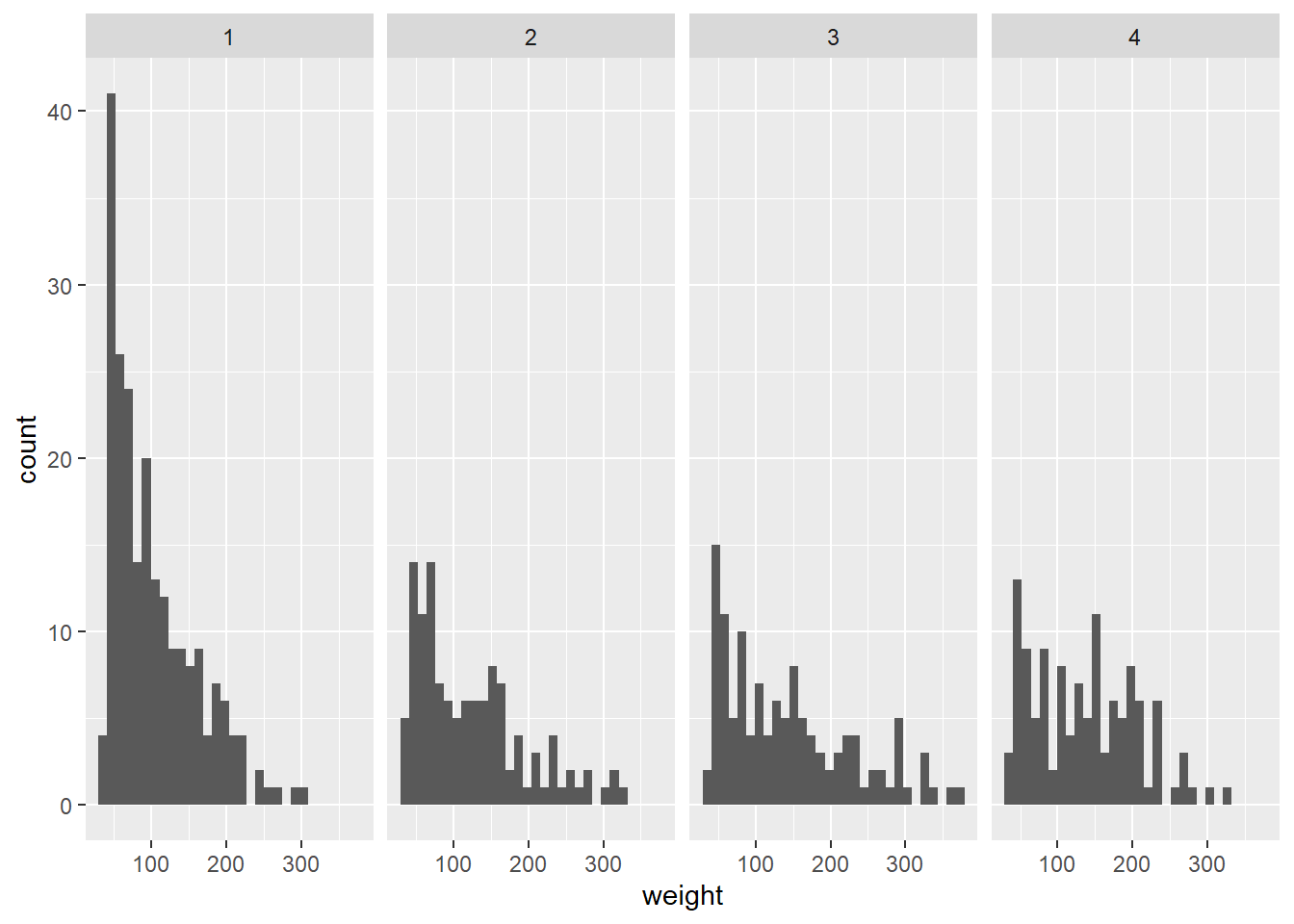
##
## Attaching package: 'dplyr'## The following objects are masked from 'package:stats':
##
## filter, lag## The following objects are masked from 'package:base':
##
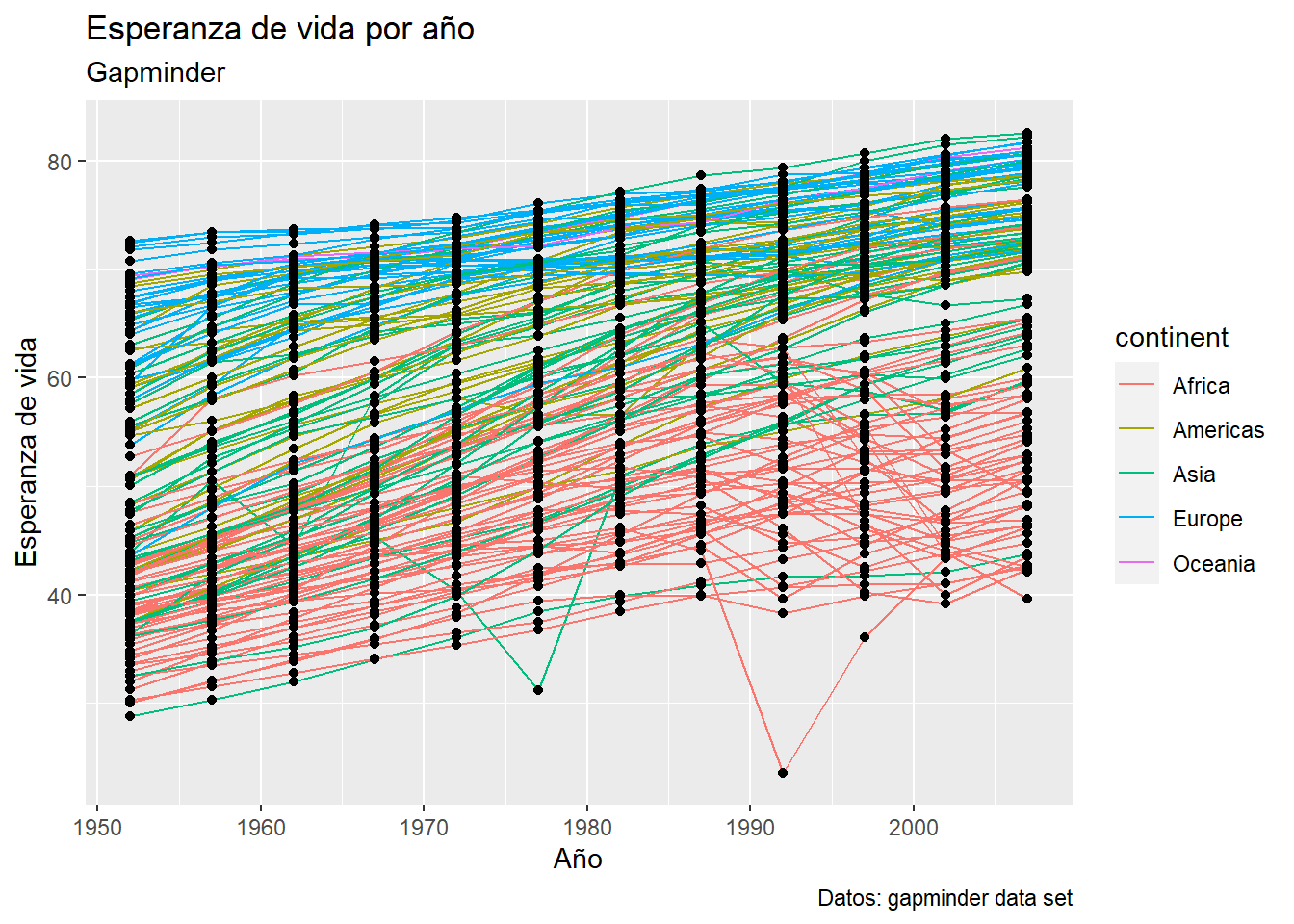
## intersect, setdiff, setequal, uniongapminder <- gapminder::gapminder
ggplot(gapminder, aes(x= gdpPercap, y = lifeExp, colour = continent)) +
geom_jitter() +
facet_grid(continent ~ year)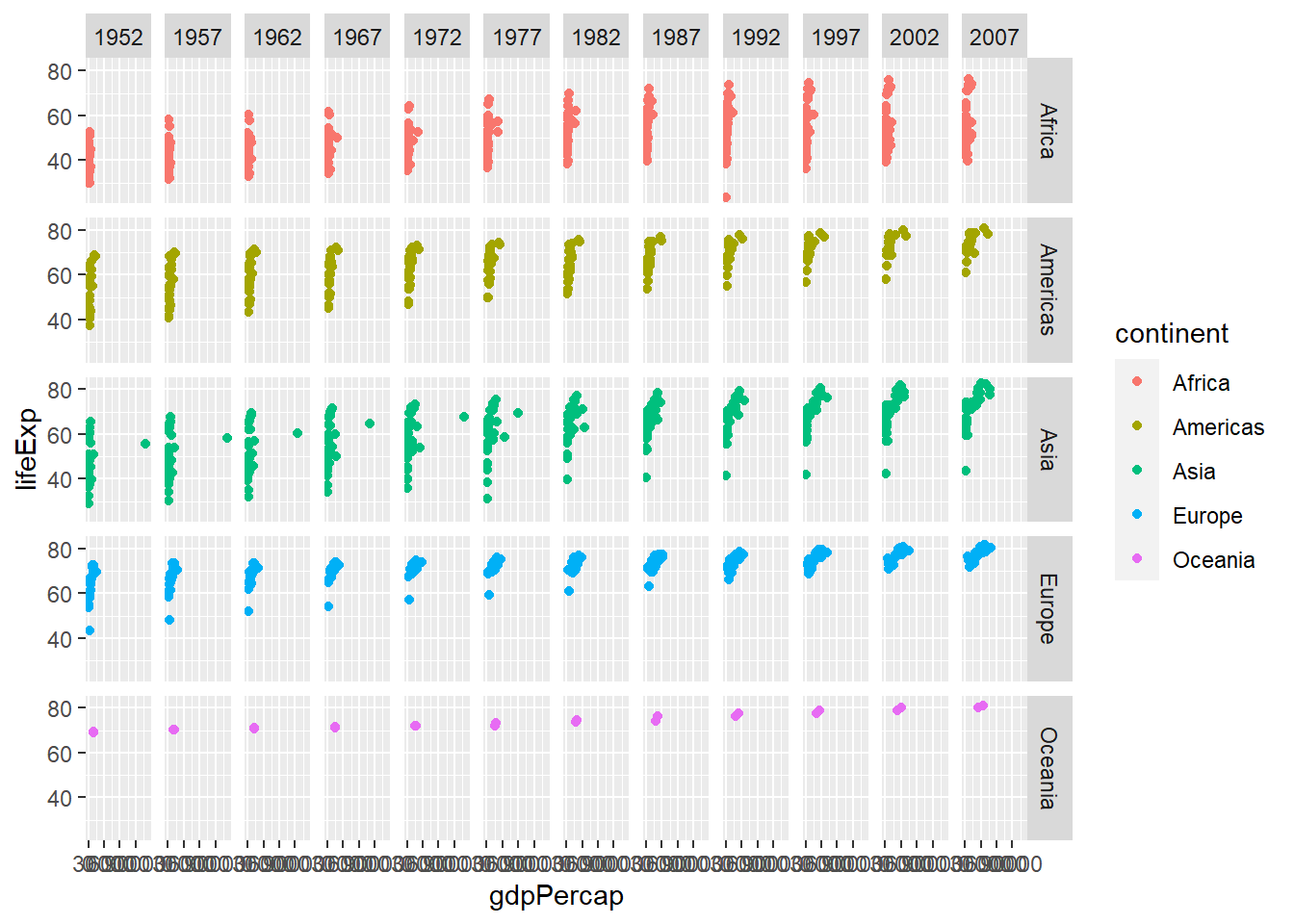
america <- gapminder %>%
filter(continent == "Americas")
ggplot(america, aes(x= year, y = lifeExp, color = continent))+
geom_line() +
facet_wrap(~country)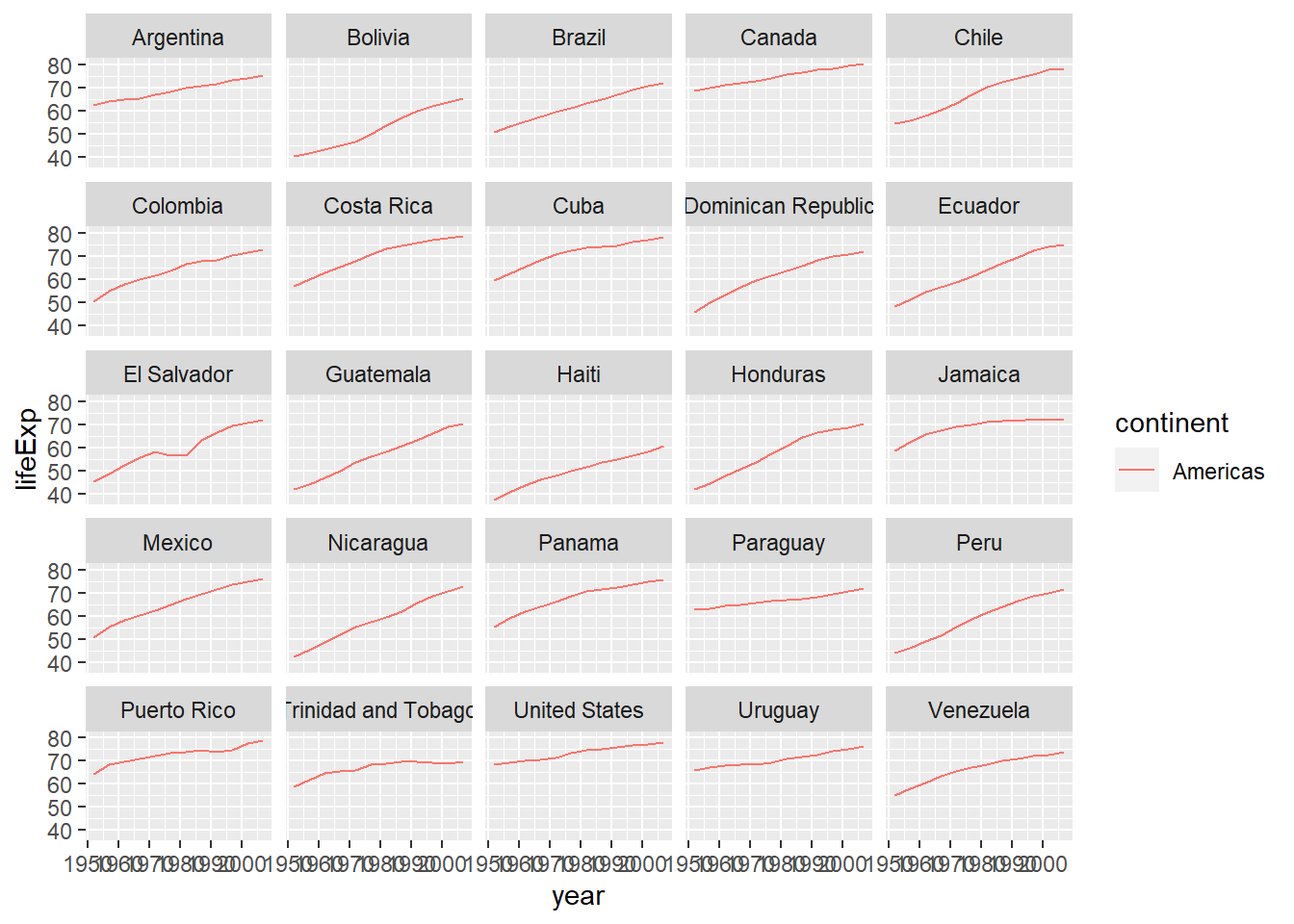
Nota: Multi panel plots con cowplot.
5.3 3D plots
Existen varios paquetes para crear gráficos en 3D, algunos de ellos son: scatterplot3d, RGL, lattice. Primero vamos a instalar el paquete scatterplot3d y cargarlo.
La forma básica de mandarlo a llamar es la siguiente:
Las variables \(y,z\) son opcionales en los casos de que \(x\) sea una fórmula, por ejemplo z~x+y*x y en el caso de que \(x\) sea una matriz con al menos 3 columanas correspondientes a las variables \(x,y\) y \(z\). Vamos a cargar la base de datos de iris.
Los parámetros de título y etiquetas de ejes funcionan como en el caso 2d. Otro parámetro que podemos cambiar es el ángulo de la perspectiva.
scatterplot3d(iris[,1:3], angle = 75,
main = "Gráfico de dispersión 3D",
xlab = "Longitud Sepal (cm)",
ylab = "Ancho Sepal (cm)",
zlab = "Longitud de Pétalo (cm)"
)Podemos colorear y poner diferentes símbolos por especies.
formas <- c(16,17,18)
formas <- formas[as.numeric(iris$Species)]
colores <- c("#FF33CC", "#CC66FF", "#33C9CC")
scatterplot3d(iris[,1:3],
pch = formas,
color = colores[iris$Species],
main = "Gráfico de dispersión 3D",
xlab = "Longitud Sepal (cm)",
ylab = "Ancho Sepal (cm)",
zlab = "Longitud de Pétalo (cm)"
)Podemos manipular la apariencia de los grid y ejes.
formas <- c(16,17,18)
formas <- formas[as.numeric(iris$Species)]
colores <- c("#FF33CC", "#CC66FF", "#33C9CC")
scatterplot3d(iris[,1:3],
pch = formas,
color = colores[iris$Species],
main = "Gráfico de dispersión 3D",
xlab = "Longitud Sepal (cm)",
ylab = "Ancho Sepal (cm)",
zlab = "Longitud de Pétalo (cm)",
grid = TRUE, box = FALSE
)En la página STHDA existe una función para personalizar los grids. Para usarla debemos descargarla y guardarla en la ruta correcta para poder llamarla en nuestro código.
# Llamar a la función
source("~/R_sites/Seminario_Estadistica/scripts/addgrids3d.R")
# Crear el plot 3d
formas <- c(16,17,18)
formas <- formas[as.numeric(iris$Species)]
colores <- c("#FF33CC", "#CC66FF", "#33C9CC")
scatterplot3d(iris[,1:3], pch = formas,
color = colores[iris$Species],
main = "Gráfico de dispersión 3D",
xlab = "Longitud Sepal (cm)",
ylab = "Ancho Sepal (cm)",
zlab = "Longitud de Pétalo (cm)",
grid = TRUE, box = FALSE
)
# Llamar a la función addgrids3d
addgrids3d(iris[, 1:3], grid = c("xy", "xz", "yz"))En esta versión los puntos se ven atrás del grid, para modificar esto primero creamos un plot vacío y después se añaden los puntos.
# Llamar a la función
source("~/R_sites/Seminario_Estadistica/scripts/addgrids3d.R")
# Configuración
formas <- c(16,17,18)
formas <- formas[as.numeric(iris$Species)]
# Crear plot vacío
plot3d <- scatterplot3d(iris[,1:3], pch = "",
grid = FALSE,
box = FALSE,
main = "Gráfico de dispersión 3D",
xlab = "Longitud Sepal (cm)",
ylab = "Ancho Sepal (cm)",
zlab = "Longitud de Pétalo (cm)"
)
# Llamar a la función addgrids3d
addgrids3d(iris[, 1:3], grid = c("xy", "xz", "yz"))
# Agregar puntos
plot3d$points3d(iris[,1:3], pch = formas)Para añadir las leyendas se puede realizar con la instrucción legend.
formas <- c(16,17,18)
formas <- formas[as.numeric(iris$Species)]
colores <- c("#FF33CC", "#CC66FF", "#33C9CC")
plot3d <-scatterplot3d(iris[,1:3], pch = formas,
color = colores[iris$Species],
main = "Gráfico de dispersión 3D",
xlab = "Longitud Sepal (cm)",
ylab = "Ancho Sepal (cm)",
zlab = "Longitud de Pétalo (cm)",
grid = TRUE
)
legend("bottom", legend = levels(iris$Species),
col = colores,
pch = c(16,17,18),
inset = -0.25, xpd = TRUE, horiz = TRUE)Una forma de tener plots interactivos es con la función plotrgl() de la librería rgl. Para eso, necesitamos primero un plot usando la librería plot3Drgl.
# Creamos el plot usando funciones de plot3D
scatter3D(iris$Sepal.Length, iris$Sepal.Width, iris$Petal.Length, bty = "g", pch = 17,
col.var = as.integer(iris$Species),
col = colores[iris$Species])
# Lo convertimos en un plot rgl interactivo
#plotrgl()La librería car cuenta con otras funciones para realizar plots 3D.
rgl.open()
# Plot 3d con plano de regresión
scatter3d(iris$Sepal.Length, iris$Sepal.Width, iris$Petal.Length)
rgl.clear()rgl.open()
# Quitar grid y colorear por especies
scatter3d(iris$Sepal.Length, iris$Sepal.Width, iris$Petal.Length,
groups = iris$Species, grid = FALSE)
rgl.clear()# Agregar un fit smooth
scatter3d(iris$Sepal.Length, iris$Sepal.Width, iris$Petal.Length,
groups = iris$Species, grid = FALSE,
fit = "smooth")# Quitar planos y agregar elipses de concentración
scatter3d(iris$Sepal.Length, iris$Sepal.Width, iris$Petal.Length,
groups = iris$Species, surface = FALSE, ellipsoid = TRUE, grid = FALSE)En la página de STHDA se encuentra un tutorial muy completo sobre plots 3D.
La función persp del paquete de R base nos ayuda a generar plots de superficies. Para usar esta función debemos indicarle los vectores de coordenadas de \(x,y\).
# Función a graficar
cono <- function(x,y){
sqrt(x^2 + y ^2)
}
# Vectores X,Y
x <- seq(-10,10, length(100))
y <- seq(-10,10, length(100))
z<- outer(x,y, cono)
# Superficie generada
persp(x,y,z)Al igual que con las funciones de R base, se pueden personalizar estos plots.
# Función a graficar
cono <- function(x,y){
sqrt(x^2 + y ^2)
}
# Vectores X,Y
x <- seq(-10,10)
y <- seq(-10,10) #length(100))
z<- outer(x,y, cono)
# Superficie generada
persp(x,y,z,
main = "Plot 3D cono",
zlab = "Altura",
col = "blue",
shade = 0.4)En la librería plot3D existe la función surf3D().
# el grid
M <- mesh(seq(0, 6*pi, length.out = 50),seq(pi/3, pi, length.out = 50))
# coordenadas auxiliares
u <- M$x ; v <- M$y
# Parametrización
x <- v * cos(u)
y <- v * sin(u)
z <- 10 * u
# Superficie
surf3D(x, y, z, colvar = z, colkey = TRUE,
box = TRUE, bty = "b", phi = 20, theta = 120)Si lo que queremos es un plot interactivo debemos usar plot3d y rglwidget.
# Grid
R <- 3; r <- 2
M <- mesh(seq(0, 2*pi,length.out=50), seq(0, pi,length.out=50))
alpha <- M$x; beta <- M$y
# Parametrización Toro
x <- (R + r*cos(alpha)) * cos(beta)
y <- (R + r*cos(alpha)) * sin(beta)
z <- r * sin(alpha)
# Plot 3d
torus <- plot3d(x, y, z, type = "l", col = "red",
cex = .3, pch = 1, main = "Toro", pch = 20)
rglwidget(elementId = "plot3drgl")Nota: Otros ejemplos los pueden encontrar en el link.