Sección 3 Manejo de Proyectos
3.1 Proyectos
Una buena practica es mantener todos nuestros trabajos en proyectos, de esta forma las variables, scripts, imágenes que generemos se quedaran asociados solo a un proyecto y no a todo el ambiente de R.
Para crear un proyecto, vamos a darle clic en File > New Project
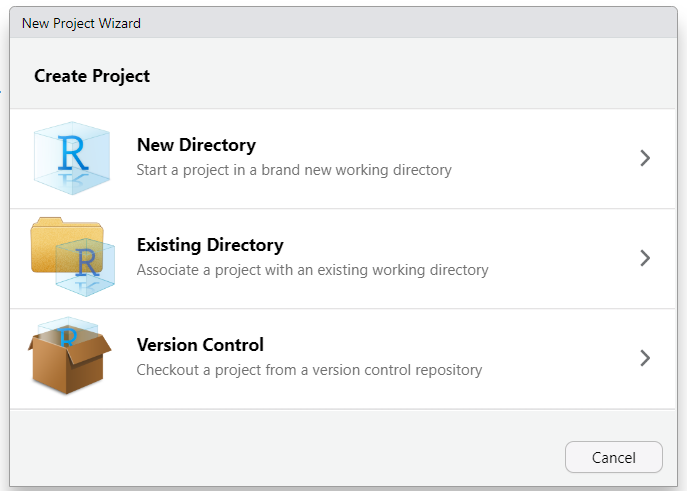
Una vez que creemos el proyecto, vamos a tener una nueva sesión de R con todos los paneles “limpios”. Si verificamos el directorio de trabajo con getwd() vamos a ver que el directorio base de nuestro proyecto es el del directorio actual de trabajo.
Vamos a crear el siguiente script de R y guardarlo con el nombre iris.R.
library(ggplot2)
data("iris")
ggplot(iris, aes(Petal.Length, Petal.Width))+
geom_point(aes(color=Species, shape= Species))
ggsave("iris.pdf")
write.csv(iris, "iris.csv")Una vez que corran todo el código en su proyecto se les generaran dos archivos, uno llamado iris.pdf y el otro iris.csv. Ahora, cierren RStudio e inspeccionen el folder que crearon. Dándole doble clic al archivo R Project, van a abrir de nuevo su proyecto y entraran todo todo tal cual lo dejaron, conservara la historia de sus comandos pero tendrán un ambiente limpio (no van a tener guardadas las variables).
Algunas recomendaciones para tener organizado un proyecto son las siguientes:
- Tener cada proyecto en su propio directorio.
- Colocar los documentos de texto asociados al proyecto en el directorio
doc. - Poner las bases de datos originales (raw data) en el directorio
datay los resultados o bases de datos procesados enresults. - Colocar los scripts y programas en el directorio
src. - Llamar a los archivos con nombres que reflejen el contenido de ellos.
Dentro de results también puedes crear carpetas con las imágenes img, resultados particulares por separado.
3.2 RMarkdown
R Markdown es una herramienta que integra escritura, código y resultados para comunicar nuestros análisis. Los documentos de R Markdown son completamente reproducibles y soportan varios tipos de archivos de salida: pdf, Word, diapositivas, html.
Los archivos de R Markdown están diseñadas para trabajar de las siguientes formas:
- Para comunicarse con los tomadores de decisiones, que quieren centrarse en resultados y no el código detrás del análisis.
- Para colaborar con otros científicos de datos que están interesados tanto en los resultados como en como se llego a ellos.
- Como un entorno en el que hacer ciencia de datos, un cuaderno de laboratorio donde están plasmadas las ideas y lo que se realizó.
La ayuda para R Markdown no se encuentra con la forma usual ?, para buscar la ayuda debes ir al menú Help > Cheat Sheets > R Markdown Cheat Sheet y Help > Cheat Sheets > R Markdown Reference Guide.
Lo primero que haremos será instalar el paquete de RMarkdown.
# Install from CRAN
#install.packages('rmarkdown')Si ya tienen alguna versión de Latex, no hacer nada, Si no tienen instalado Latex, entonces instalar alguna distribución MiKTeX, MacTeX, and TeX Live o TinyTex
#install.packages('tinytex')
#tinytex::install_tinytex()
# to uninstall TinyTeX, run
# tinytex::uninstall_tinytex()Para abrir un documento R Markdown, vamos a File > New File > R Markdown.
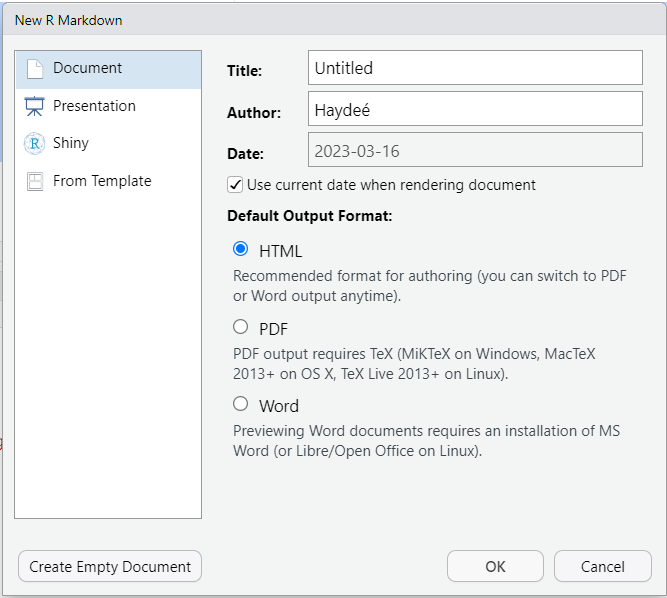
El chunk inicial de un documento RMarkdown incluye el tipo de documento que generará, el autor, título, fecha.
Estos campos se pueden borrar o cambiar. Por default, nos crea un documento lleno con un ejemplo de como se usan los documentos.
Los chunks de R los podemos correr uno por uno o podemos usar la opción Knit, esto último nos va a generar el archivo completo.
3.2.1 Formato de texto con R Markdown
El texto en los archivos .Rmd se escribe usando Markdown el cual fue diseñado para que fuera fácil de leer y escribir. Podemos darle formato a las letras en cursiva o negrita como sigue:
# Cursiva
# *italic* o _italic_
# Negrita
# **bold** o __bold__
# superscript^2^
# Tachado
# ~~tachado~~Los títulos se señalan con # siguiendo el siguiente orden:
# # Título principal
# ## Subtítulo
# ### Título de 3er nivelPara escribir listas usamos la sintaxis siguiente:
# * item 1
# * item 2
# * item 2a
# * item 2b
# 1. item 1
# 2. item 2
# a. item 2aPara insertar links e imágenes usamos lo siguiente:
# Links:
# [link](dirección)
# Imágenes:
# Una forma de generar tablas es la siguiente:
# First Header | Second Header
# ------------- | -------------
# Content Cell | Content Cell
# Content Cell | Content Cell3.2.2 Código en R
Existen dos tipos de código en Rmarkdown, código en línea y chunks. La sintaxis para escribir código R en línea es
` r R_CODE `. Por ejemplo, supongamos que queremos calcular la suma de \(1+1\), entonces escribimos 2.
Para los chunks, veamos lo siguiente.
Leemos la base de datos y la asignamos a la variable gapminder. Vamos a agregar un nombre a nuestro chunk y los atributos message=FALSE, warning=FALSE para que no se desplieglen los mensajes de error o los warnings si es que existen.
Vamos a realizar un gráfico de dispersión para darnos una idea de como están los datos en nuestra base de datos gapminder, las variables que usaremos serán gdpPercap y lifeExp.
library(ggplot2)
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) +
geom_point()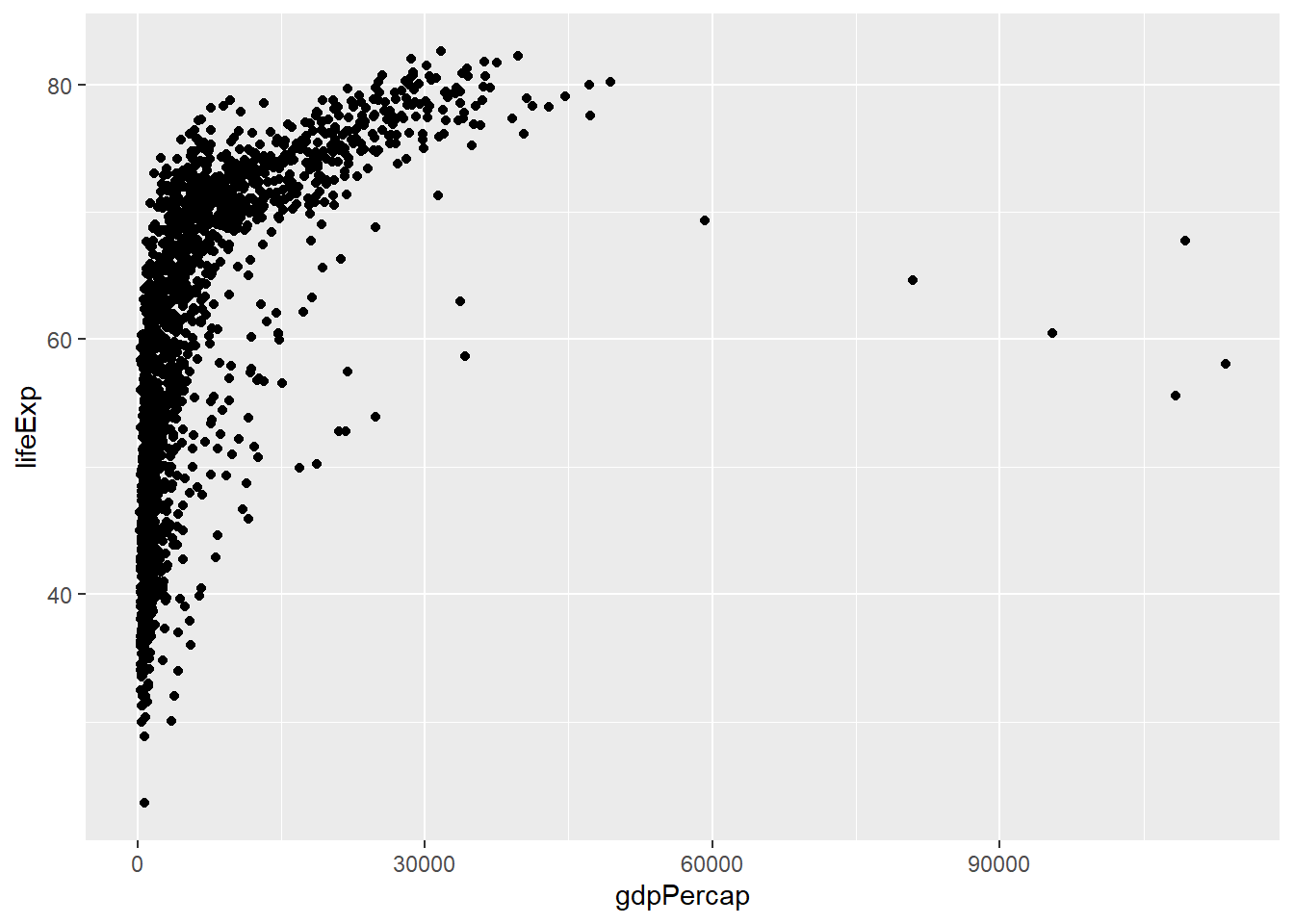
Figure 3.1: Gapminder dataset
Atributos del chunk:
fig.cap: nos sirve para darle un título a nuestro gráfico. Para hacer referencia a dicho gráfico más adelante, se usa la sintaxis\ref{fig:nombre}.fig.width,fig.height: nos indica el tamaño real de la figura.out.width: nos indica el tamaño del output de la figura.fig.align: nos indica que la posición de la figura.
Para mostrar dos plots juntos, usamos fig.show='hold'.
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) +
geom_point()
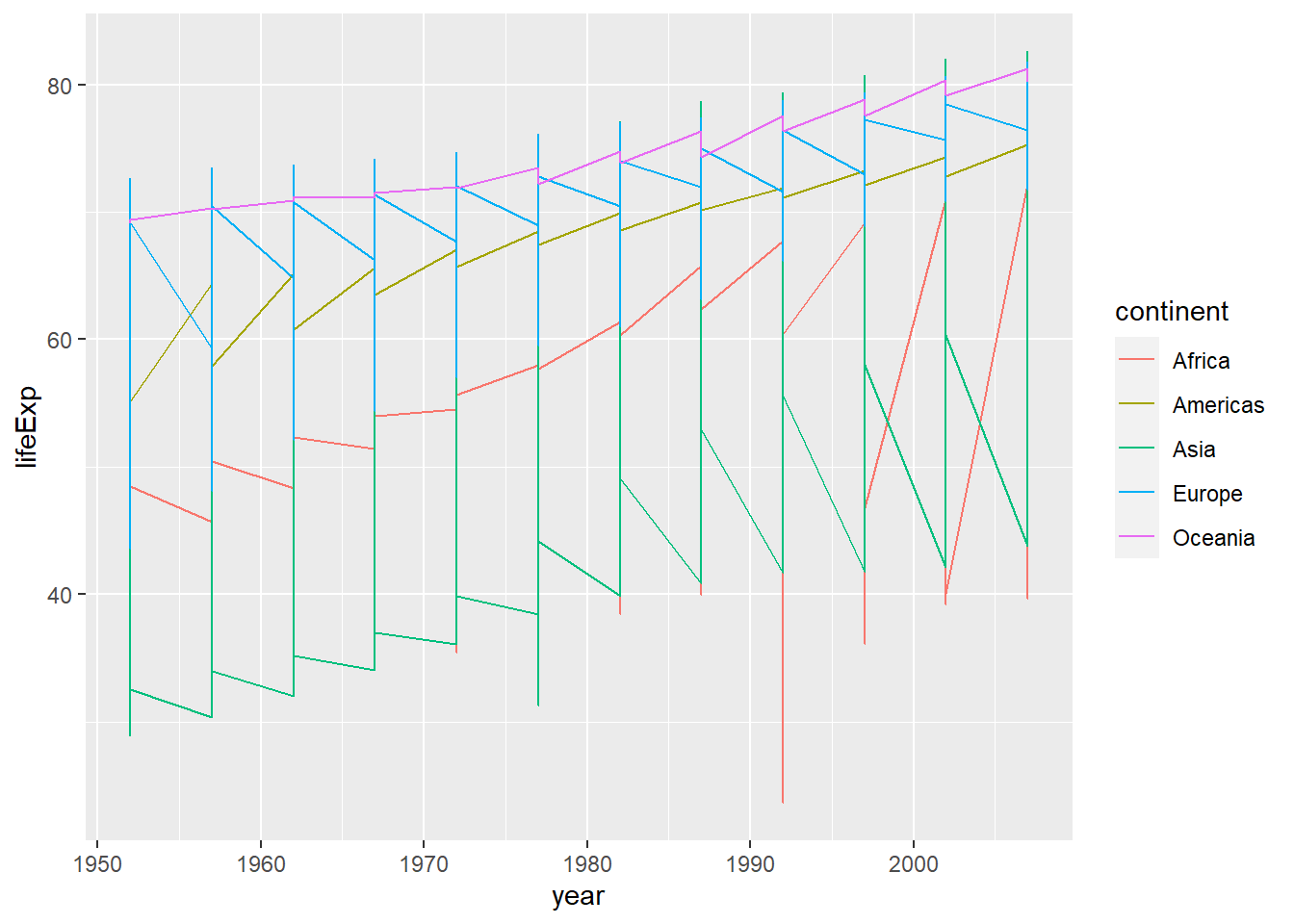
ggplot(data=gapminder, aes(x=year, y=lifeExp, color=continent))+
geom_line()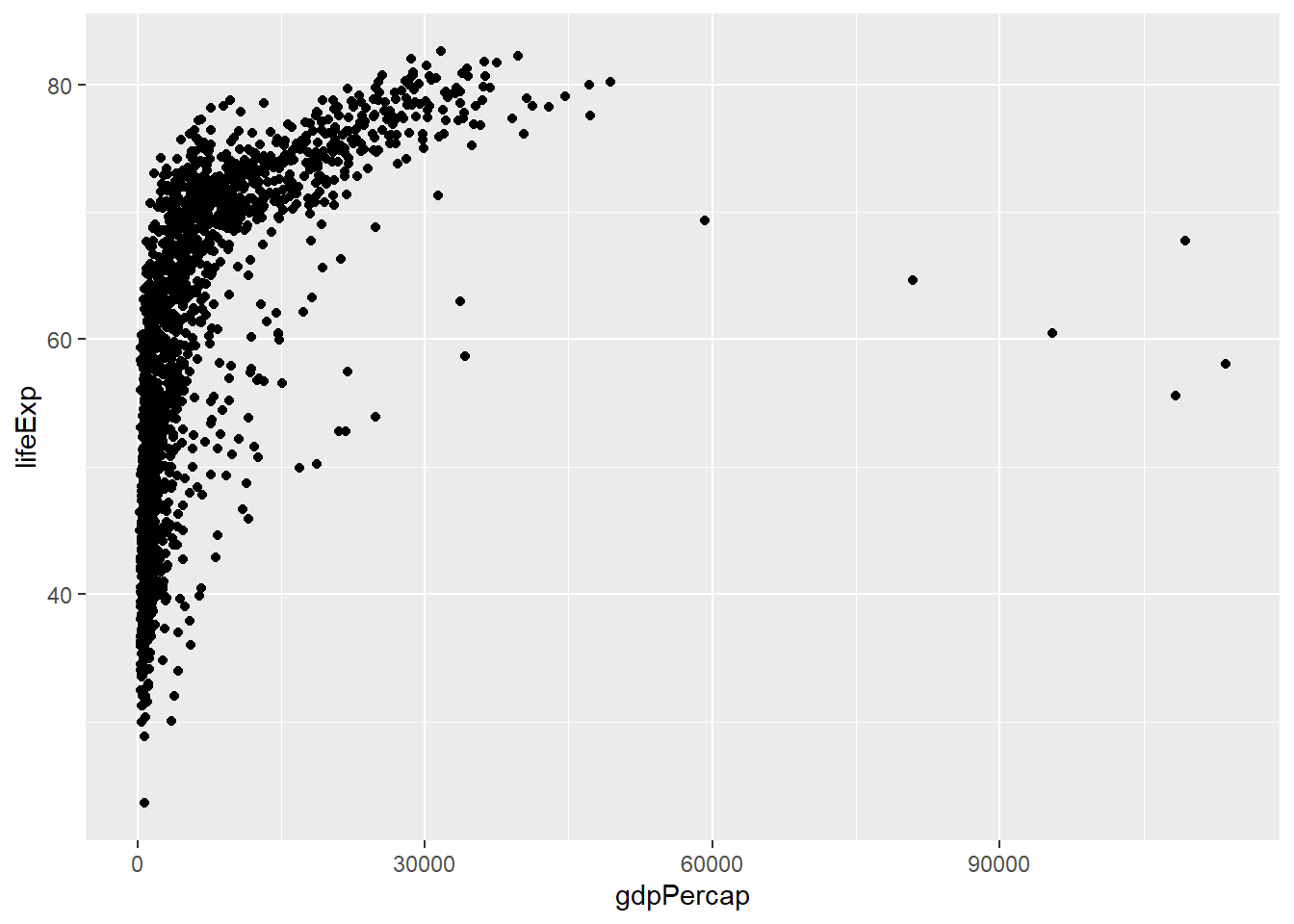
En los chunks, podemos modificar su queremos que aparezca el código de R o no cambiando la opción de include=TRUE o include=FALSE.
calcGDP <- function(dat, year=NULL, country=NULL) {
if(!is.null(year)) {
dat <- dat[dat$year %in% year, ]
}
if (!is.null(country)) {
dat <- dat[dat$country %in% country,]
}
gdp <- dat$pop * dat$gdpPercap
new <- cbind(dat, gdp=gdp)
return(new)
}Vamos a usar la función con los parámetros year=2007 y country=Australia.
calcGDP(gapminder, year=2007, country="Australia")## country continent year lifeExp pop gdpPercap gdp
## 1 Australia Oceania 2007 81.235 20434176 34435.37 703658358894También podemos definir ciertas variables en los chunks y después mandarlas a llamar dentro del texto.
Vamos a calcular la media de la columna dgp de Africa, la vamos a asignar a una variable para después poderla llamar en cualquier parte del texto.
withGDP <- calcGDP(gapminder)
media_Africa_gdp <- mean(withGDP[withGDP$continent == "Africa", "gdp"])La media es 2.0904783^{10}.
Por default, R Markdown imprime las tablas en la misma forma que las vemos en la consola. Si queremos darle un formarto a estas tablas podemos usar el paquete kable o usar la opción knit::kable.
knitr::kable(
gapminder[1:5, ],
caption = "Una tabla con knit kable."
)| country | continent | year | lifeExp | pop | gdpPercap |
|---|---|---|---|---|---|
| Afghanistan | Asia | 1952 | 28.801 | 8425333 | 779.4453 |
| Afghanistan | Asia | 1957 | 30.332 | 9240934 | 820.8530 |
| Afghanistan | Asia | 1962 | 31.997 | 10267083 | 853.1007 |
| Afghanistan | Asia | 1967 | 34.020 | 11537966 | 836.1971 |
| Afghanistan | Asia | 1972 | 36.088 | 13079460 | 739.9811 |
También podemos usar todos los comandos de latex para insertar figuras, teoremas, ecuaciones, listas, tablas, etc.
3.3 Proyectos colaborativos (Git + GitHub)
Generalmente cuando comenzamos un proyecto, el primer borrador no es el final y queremos una forma de poder llevar un registro de los cambios sobre el tiempo. Cuando colaboramos con otras personas, una forma de compartir el trabajo y hacer ediciones es enviarnos los archivos por email o manternerlos en alguna carpeta que todos tengamos acceso, sin embargo, si a alguien se le olvida avisarnos que hizo cambios o no actualiza los archivos vamos a estar trabajando sobre archivos diferentes y probablemente tengamos perdida de información o que lo que alguien realizo no nos coincide a nosotros.
Git es un sistema de control de versiones creado para desarrolladores para colaborar en grandes proyectos. Git nos ayuda a llevar un registro de los cambios que se han realizado y quien ha realizado los cambios, además guarda las versiones anteriores y en caso de algún conflicto con versiones recientes siempre es posible regresar a alguna versión previa donde todo funcionaba correctamente. Cada proyecto se guarda en un repositorio que incluye todos los archivos que forman parte del proyecto, esto puede ser bases de datos, reportes, imágenes, scripts, etc.
Git se puede usar localmente pero si se requiere compartir todo con colaboradores, entonces se debe almacenar en la web para que otros lo puedan descargar. Los repositorios pueden ser públicos o privados. GitHub se ha convertido en uno de los mayores hospederos de repositorios de Git que además incluye algunas otras características útiles.
3.3.1 Instalar Git
Instalar Git for Windows:
- Configurar su PATH: seleccionar
Git from the command line and also from 3rd-party software. - RStudio prefiere que Git esté instalado en
C:/Program Filesque suele ser el de default.
3.3.2 Configurar Git
Primero debemos asegurarnos que tenemos instalados los paquetes adecuados:
# install.packages(c("usethis", "gitcreds", "gh"))Ahora debemos identificarnos con nuestras credenciales de GitHub.
#usethis::use_git_config(user.name = "Usuario GitHub", user.email = "email de la cuenta de GitHub")Para poder subir los cambios a GitHub, necesitamos autenticarnos, es decir, probar que somos los dueños de la cuenta de GitHub.
3.3.3 Cache credentials for HTTPS
Con este método, se pueden clonar los repositorios usando el url que sale en HTTPS. Para este, necesitamos un personal access token (PATH).
Para obtener el PATH, vamos a correr lo siguiente en la consola de R:
usethis::create_github_token(
scopes = c("repo", "user", "gist", "workflow"),
description = "alguna descripcion",
host = "https://github/user/"
)Esta función nos abrirá un navegador para generar el PATH:
- Dar una descripción al PATH: por ejemplo “Curso Estadistica”.
- Cambiar la fecha de vencimiento: la fecha puede ser por cierta cantidad de días o sin fecha de vencimiento.
- Dejar las demas opciones default y seleccionar
Generar token. Este token deben de guardarlo en algún lugar ya que no es posible verlo de nuevo una vez que cierren la página.
Si olvidan su PATH es posible generar otro.}
Para guardar el PATH, ejecuten el siguiente código en la consola de RStudio:
gitcreds::gitcreds_set(url = "https://github.user")Cuando se habrá la ventana les pedirá pegar su PATH en la consola y presionar enter. De está forma las credenciales ya quedarán guardadas en su computadora.
Para confirmar que su PATH si se guardo correctamente, correremos el siguiente código:
gh::gh_whoami(.api_url = "https://github.user")
usethis::git_sitrep()Lo que deberán ver son sus datos de su cuenta de GitHub.
3.3.4 Trabajo colaborativo en GitHub
Una de las ventajas de usar esta herramienta es la posibilidad de tener control sobre los cambios que se hacen en trabajos colaborativos ya sea de manera sincrónica o asíncrona. Para ello, es conveniente seguir pautas que aseguren los mejores resultados, así que a continuación se indican los pasos a seguir para trabajar localmente una vez que se ha obtenido acceso al repositorio principal. Ya que nos interesa incluir código LaTeX, recomendamos visitar este libro donde se recomienda instalar Miktex (u otra distribución dependiendo de su sistema operativo) y autorizar la instalación de paquetes al vuelo (on the fly) para que no haya errores de compilación.
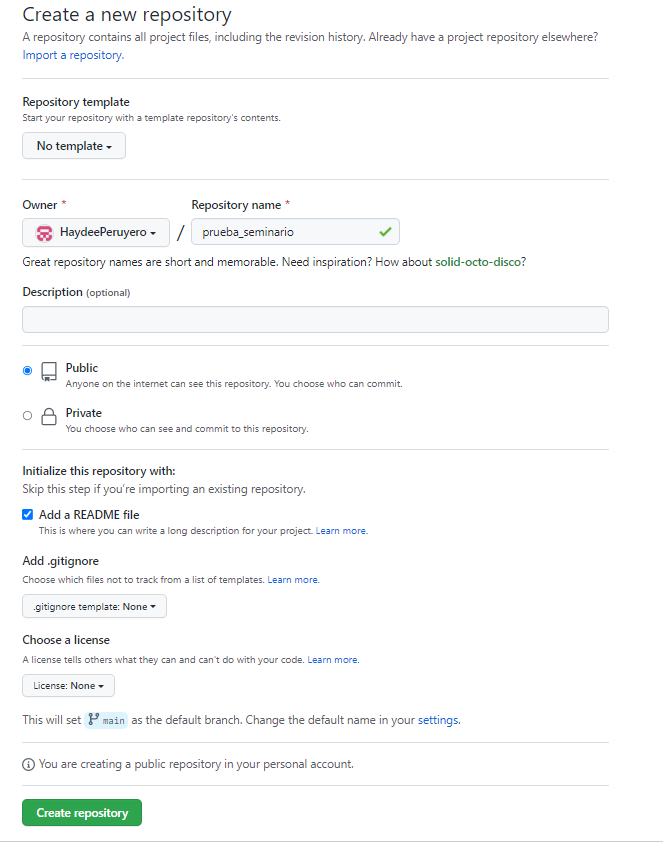
3.3.4.1 Crear repositorio en GitHub
Abrimos nuestra cuenta de GitHub y damos clic en New:
3.3.4.2 Clonar repositorio
En primer lugar clonaremos el repositorio para trabajar de manera local.
- Abrir RStudio.
- Luego, en el menú
Filehacer click enNew Project. - En la ventana que se abre, hacer click en
Version Control.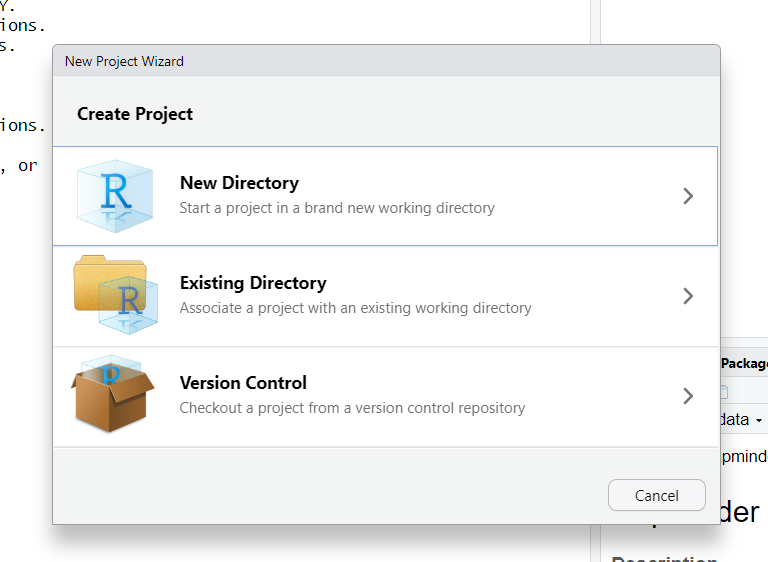
- Después hacemos click en
Git.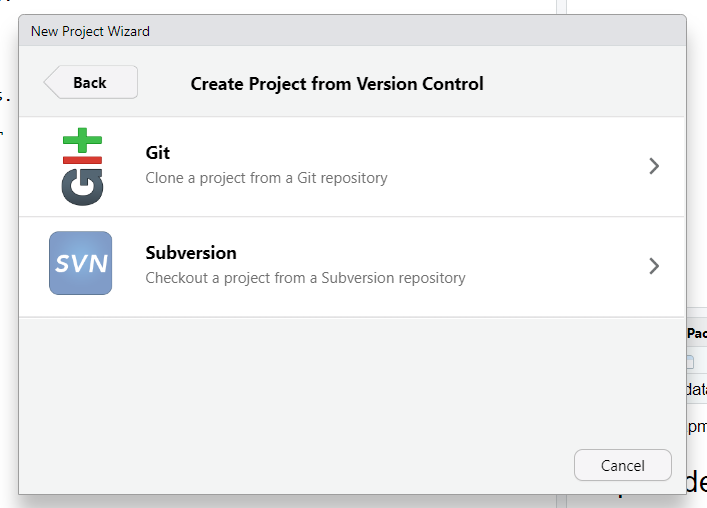
- Copiar la url del repositorio que deseamos clonar.
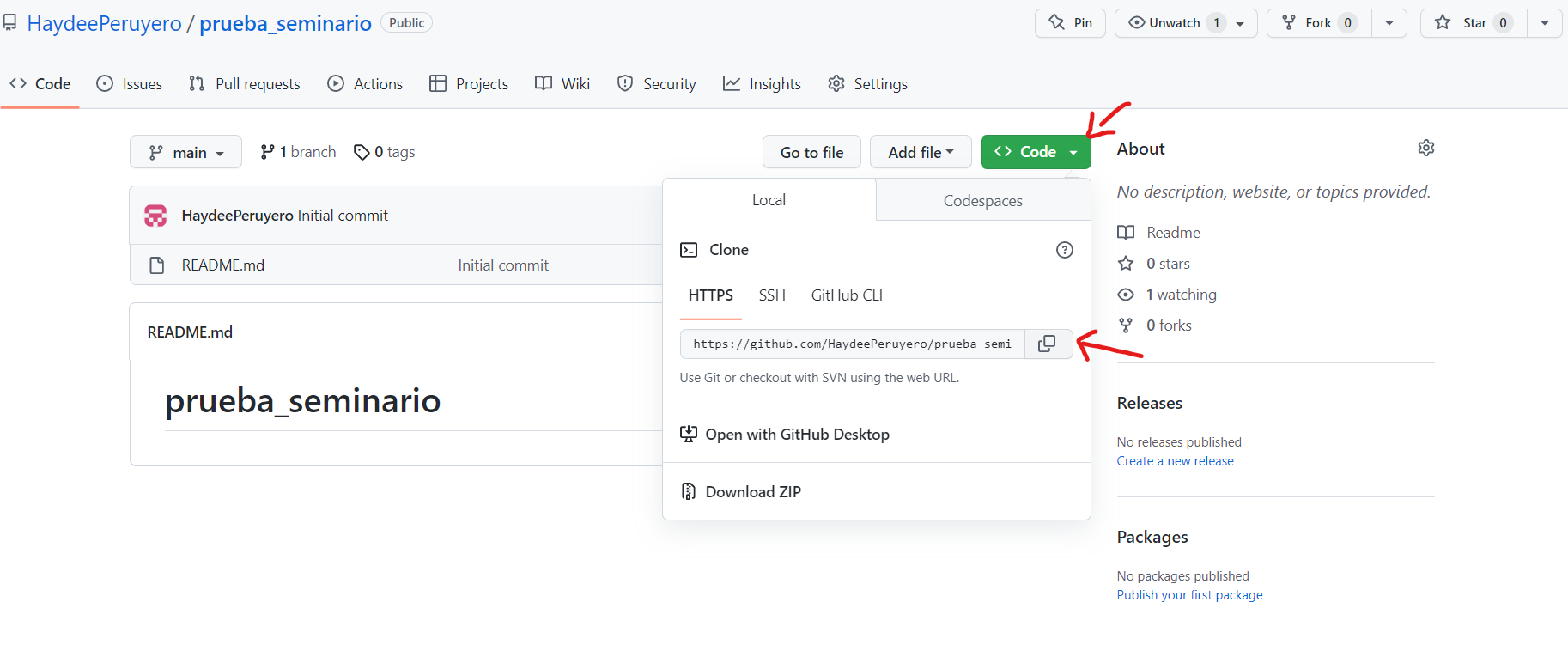
- Pegar la url en la ventana de RStudio. Cambiar el nombre del directorio (si así lo deseamos), elegir la carpeta donde se va a guardar el clon del repositorio. Marcar
Open in new sessionpara que el proyecto utilice su propia sesión deR.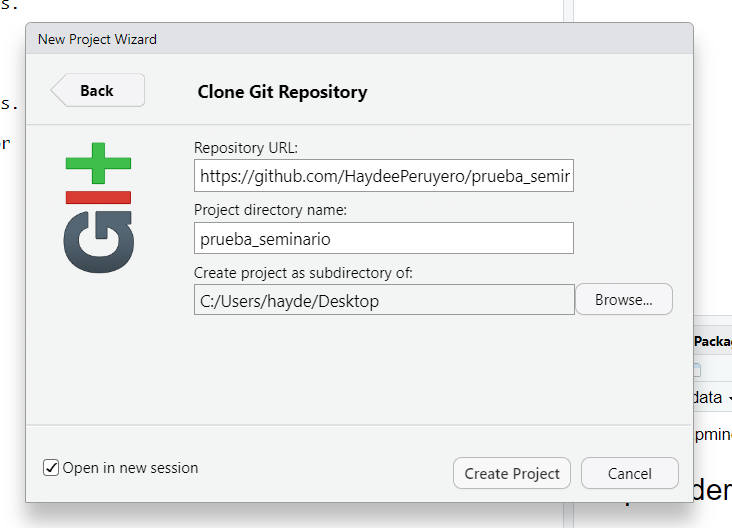 Y presionar en
Y presionar en Create Projectpara que se cree el nuevo proyecto.
Una vez realizado lo anterior ya se puede trabajar dentro del proyecto. Por ahora, la explicación pasará a qué hacer cuando abrimos nuevamente un proyecto ya clonado, posteriormente estableceremos el flujo de trabajo.
3.3.4.3 Abrir proyecto ya creado
En esta parte supondremos que ya tenemos nuestro directorio creado.
- Abrir RStudio.
- En el menú
Filehacer click enOpen project in New Session... - En la ventana que se abre ir al directorio donde se encuentra guardado el proyecto y abrir el archivo
prueba.Rproj.
Como recomendación, siempre trabaje en una nueva sesión. A partir de aquí ya puede continuar con su flujo de trabajo.
3.3.4.4 Flujo de trabajo general
Para esta parte supondremos que ya tiene abierto el proyecto en el cual va a trabajar. Por ahora, el flujo utiliza la terminal de RStudio (y si hay una rama local, entonces usaremos la interfaz gráfica en dos ocasiones)
3.3.4.5 Usando la terminal
En su IDE (entorno de desarrollo integrado) identifique Terminal, no la confunda con Console.
- En
Terminalejecutegit pullpara descargar la última versión disponible del repositorio. Esto asegura que se dispone de la última versión disponible del repositorio.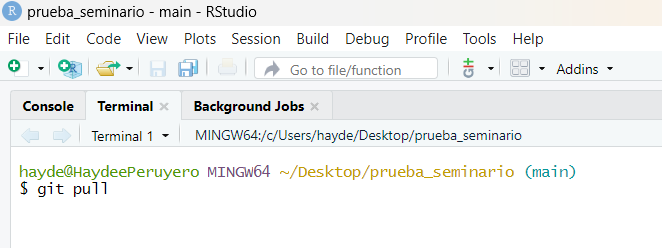
- Este paso no es necesario, pero podría evitarle problemas en caso de que haya un error catastrófico en la programación que arruine sus avances, aunque lo podría hacer desde
Terminal, es preferible hacerlo con la interfaz. Identificamos la pestañaGity hacemos click sobre el símbolo de color morado.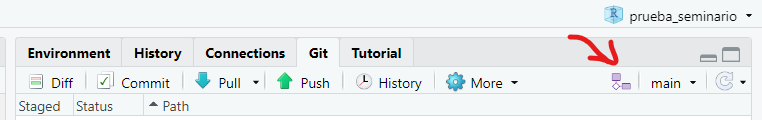 En la ventana que se abre, en
En la ventana que se abre, en Branch Name:escribir el nombre de la rama local que desee. Como sugerencia escriba un nombre corto, que no lleve acentos ni símbolos. EnRemote:elija(None)para crear una rama local (que solo existirá en su equipo). Finalmente dé click en Create.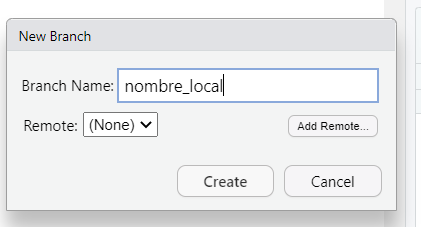
Realizamos todos los cambios que necesitemos.
A continuación vamos a guardar en el repositorio los cambios que hemos realizado.
- En
Terminalejecutamosgit add .para guardar todos los cambios (se puede hacer archivo por archivo, pero en esta ocasión agregamos todos en un solo paso sin hacer distinción, por lo cual lo invitamos a que considere si esto le conviene).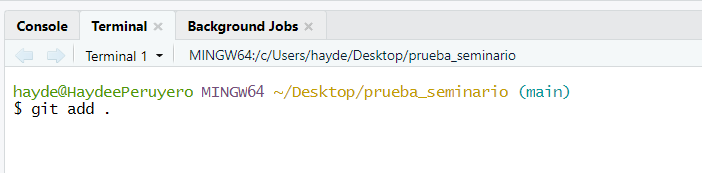
- En
Terminalejecutamosgit commit -m "mensaje de ejemplo", dondemensaje de ejemplose refiere a una pequeña descripción de los cambios realizados.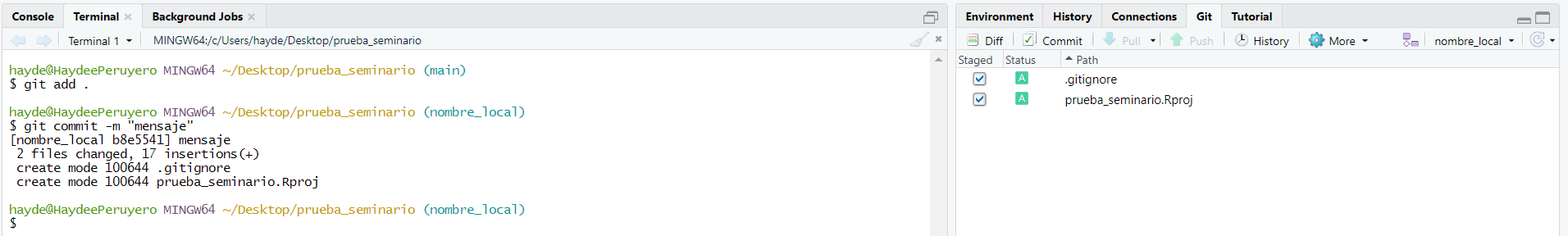
- Este paso se realiza si realizó el paso 2., en caso contrario, pase al siguiente punto.
- Junto al nombre de nuestra rama local
rama-localen la pestaña Git aparece un triángulo que apunta hacia abajo, lo pinchamos y cambiamos a la rama principalmain. Esto nos abrirá un mensaje que, si no hay errores, indicará que hemos cambiado a la rama principal.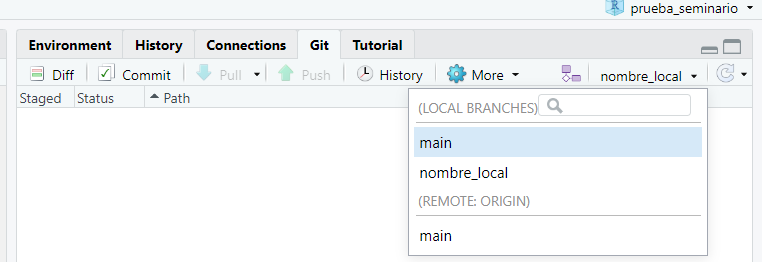
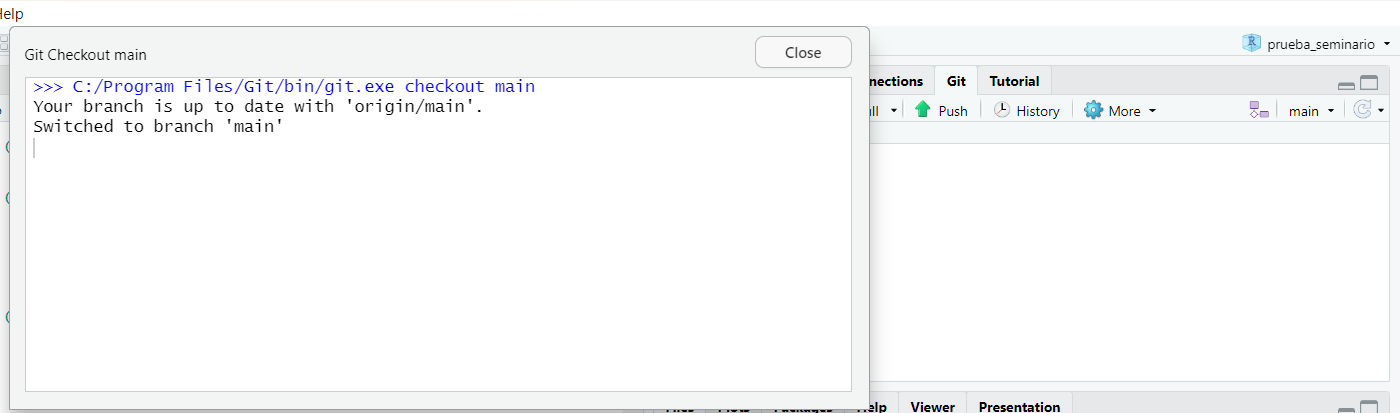
- A continuación, en
Terminalejecutamosgit pullpara llamar la versión más reciente del repositorio (podría haber cambios por parte de alguno de los colaboradores). Note que esto se está realizando en la ramamain. - Luego, en
Terminalejecutargit merge rama-local, donde recordamos que en el paso 2, como ejemplo, llamé a mi rama local comorama-local. Esto sirve para combinar los cambios que realizamos en la rama local a nuestra rama principal. - En caso necesario, si hubo cambios adicionales, se vuelve a hacer commit ejecutando
git add .ygit commit -m "mensaje"enTerminal.
Una vez que se actualizaron los cambios, debemos enviarlos a GitHub, para ello ejecutamos
git pushenTerminal.Este paso se realiza si realizó el paso 2. Ejecutamos en
Terminalel comandogit branch -d rama-localpara eliminar nuestra rama local.Para concluir, cerramos el proyecto desde
File -> Close Projectantes de cerrar RStudio para evitar errores al cargar la interfaz en una próxima ocasión.
Atención: Hay manera de incluir Python dentro de su proyecto. Debe tener instalado Python en su equipo y utilizar la librería reticulate. Vea aquí por ejemplo. De hecho, no es el único lenguaje posible.